

植物学报 ›› 2023, Vol. 58 ›› Issue (5): 743-749.DOI: 10.11983/CBB22183 cstr: 32102.14.CBB22183
田传玉1,2, 方妍力1,2, 沈晴3, 王宏杰1,2, 陈析丰1, 郭威1, 赵开军2, 王春连2,4( ), 纪志远2(
), 纪志远2( )
)
收稿日期:2022-08-03
接受日期:2023-02-09
出版日期:2023-09-01
发布日期:2023-09-21
通讯作者:
*E-mail: wangchunlian@caas.cn;jizhiyuan@caas.cn
作者简介:第一联系人:† 共同第一作者。
基金资助:
Tian Chuanyu1,2, Fang Yanli1,2, Shen Qing3, Wang Hongjie1,2, Chen Xifeng1, Guo Wei1, Zhao Kaijun2, Wang Chunlian2,4( ), Ji Zhiyuan2(
), Ji Zhiyuan2( )
)
Received:2022-08-03
Accepted:2023-02-09
Online:2023-09-01
Published:2023-09-21
Contact:
*E-mail: wangchunlian@caas.cn;jizhiyuan@caas.cn
About author:First author contact:† These authors contributed equally to this paper.
摘要: 种植抗病品种一直是防控水稻白叶枯病(bacterial blight, BB)最有效的措施。近年来, 白叶枯病在我国多地呈现“老病新发”态势。为查明近期白叶枯病成灾的原因, 2019-2021年间, 在南方8省(海南、云南、广西、广东、福建、湖南、浙江和江苏)病害重发生田块采集叶片, 分离获得野生白叶枯病菌(Xanthomonas oryzae pv. oryzae, Xoo)。通过对主效毒性因子基因型tale (transcription activator-like effectors)进行Southern杂交检测, 将新分离的97株Xoo菌株划分为10个基因型, 其中基因型V是优势种群代表。选取各基因型的代表菌株, 剪叶接种携带主要抗病基因(R gene)的水稻(Oryza sativa)品种, 毒力测试结果显示, Xa3和Xa4等传统抗病基因对田间大部分菌株已经丧失抗性, Xa7和Xa23等优异抗病基因对白叶枯病仍具有广谱抗性。研究结果表明, 近期“老病新发”的主要原因可能是水稻新品种选育过程中忽视了优异抗白叶枯病基因资源的引入, 挖掘与利用优异抗病基因资源仍是防控白叶枯病最理想的途径。
田传玉, 方妍力, 沈晴, 王宏杰, 陈析丰, 郭威, 赵开军, 王春连, 纪志远. 2019-2021年我国南方稻区白叶枯病菌的毒力与遗传多样性调查研究. 植物学报, 2023, 58(5): 743-749.
Tian Chuanyu, Fang Yanli, Shen Qing, Wang Hongjie, Chen Xifeng, Guo Wei, Zhao Kaijun, Wang Chunlian, Ji Zhiyuan. Genotypic Diversity and Pathogenisity of Xanthomonas oryzae pv. oryzae Isolated from Southern China in 2019-2021. Chinese Bulletin of Botany, 2023, 58(5): 743-749.
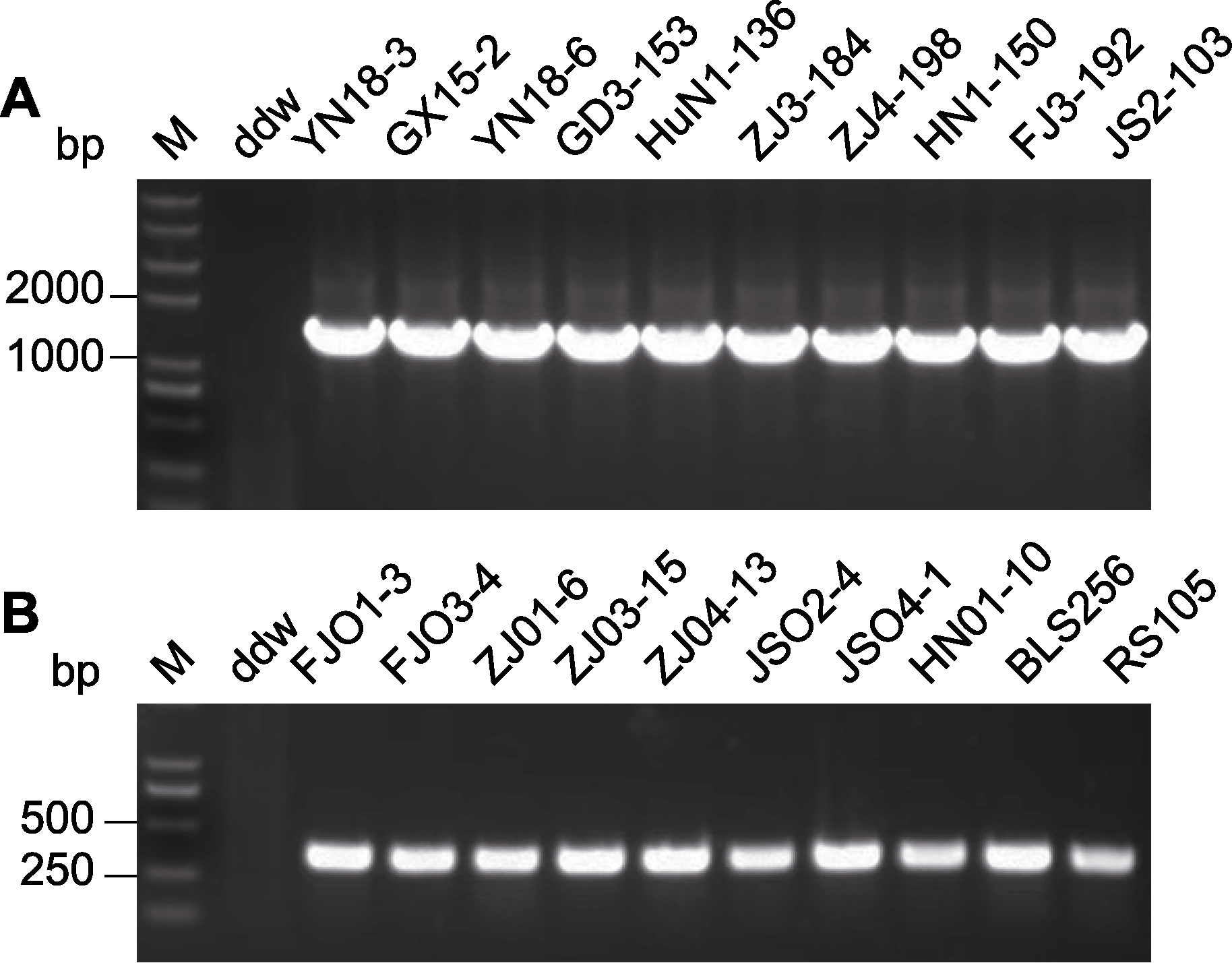
图1 2019-2021年我国南方稻区感病叶片上分离获得的水稻黄单胞菌株PCR检测 (A) 白叶枯病菌的特异性PCR检测; (B) 细菌性条斑病菌的特异性PCR检测。M: 分子量标准; ddw: 空白对照
Figure 1 PCR-based detection of Xanthomonas oryzae strains isolated from diseased leaves of Southern China in 2019-2021 (A) Specific PCR detection from Xoo strains; (B) Specific PCR detection from Xoc strains. M: Marker; ddw: Control
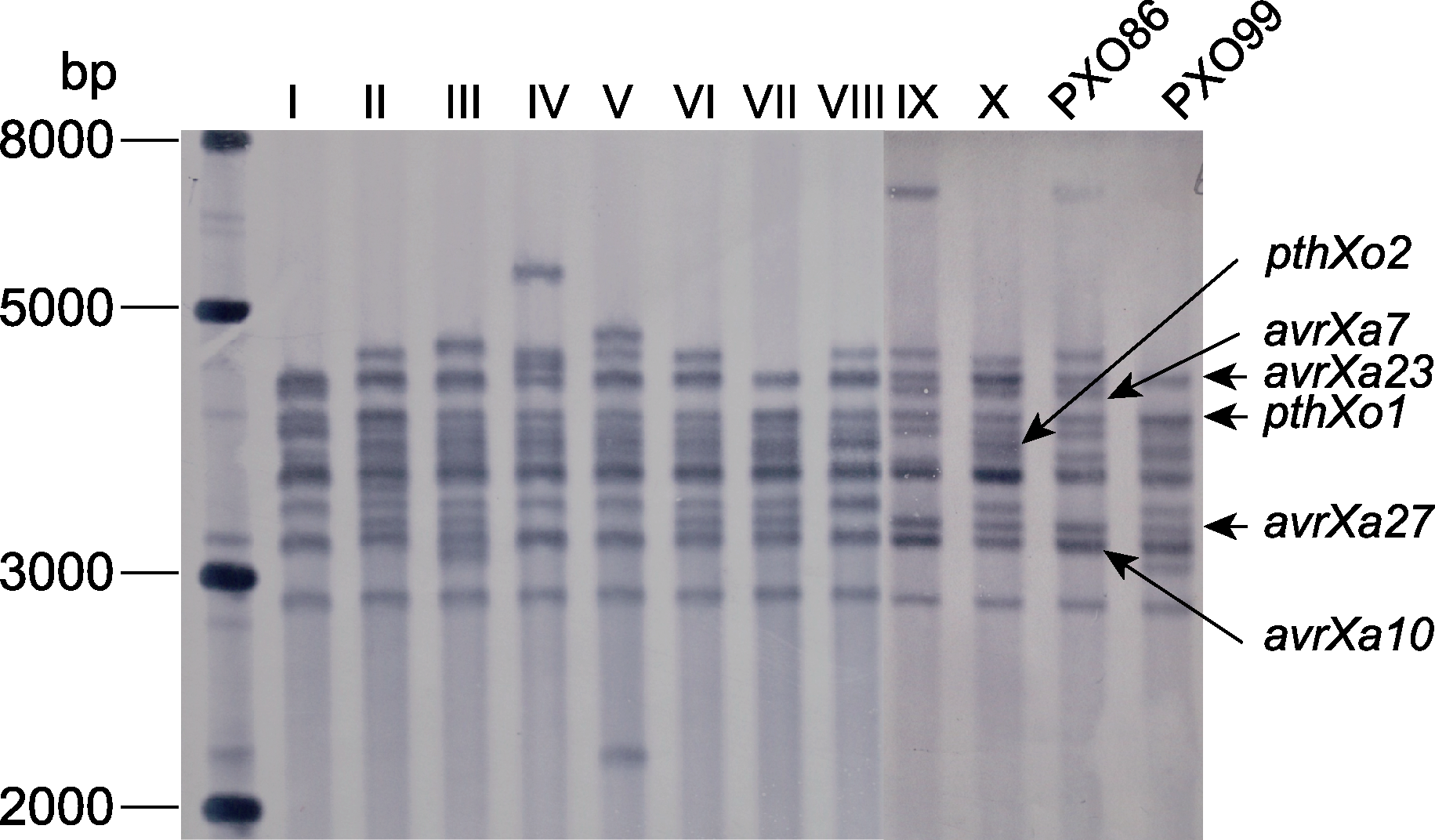
图2 2019-2021年我国南方稻区分离获得的Xoo菌株tale基因Southern杂交检测 I-X: 分离菌株代表基因型; PXO86: 菲律宾2号小种; PXO99: 菲律宾6号小种
Figure 2 Southern blot analysis of tale genes of Xoo strains collected from Southern China in 2019-2021 I-X: Genotypes of isolated strains in this study; PXO86: Philippine race 2; PXO99: Philippine race 6
| tale genotype | Representative strains | Distribution | Number of strains | Frequency (%) | Putative tale gene |
|---|---|---|---|---|---|
| I | YN18-3 | Yunnan, Guangxi | 4 | 4.12 | pthXo1, avrXa7, avrXa23 |
| II | GX15-2, YN17-8 | Yunnan, Guangxi | 2 | 2.06 | pthXo1, pthXo2, avrXa23, avrXa27 |
| III | YN17-2, YN18-6 | Yunnan, Guangdong | 8 | 8.25 | pthXo1, pthXo2, avrXa10, avrXa23, avrXa27 |
| IV | GX15-4, GD3-153 | Guangxi, Guangdong, Yunnan | 18 | 18.56 | pthXo1, pthXo2, avrXa23 |
| V | ZJ1-121, HuN1-136 | Zhejiang, Hunan, Jiangsu | 27 | 27.84 | pthXo1, pthXo2, avrXa23 |
| VI | ZJ3-184 | Zhejiang, Guangdong | 5 | 5.15 | pthXo1, pthXo2, avrXa23, avrXa27 |
| VII | ZJ4-198 | Zhejiang, Jiangsu | 4 | 4.12 | pthXo1, pthXo2, avrXa23, avrXa27 |
| VIII | GD1-105, HN1-150 | Guangdong, Hainan, Zhejiang, Guangxi | 22 | 22.68 | pthXo1, pthXo2, avrXa23 |
| IX | FJ3-192 | Fujian, Guangxi | 4 | 4.12 | pthXo1, avrXa7, avrXa10, avrXa23, avrXa27 |
| X | JS2-103 | Jiangsu, Zhejiang | 3 | 3.09 | pthXo1, pthXo2, avrXa7, avrXa10, avrXa23, avrXa27 |
表1 2019-2021年我国南方稻区分离获得的Xoo菌株tale基因型
Table 1 tale genotypes of Xoo strains collected from Southern China in 2019-2021
| tale genotype | Representative strains | Distribution | Number of strains | Frequency (%) | Putative tale gene |
|---|---|---|---|---|---|
| I | YN18-3 | Yunnan, Guangxi | 4 | 4.12 | pthXo1, avrXa7, avrXa23 |
| II | GX15-2, YN17-8 | Yunnan, Guangxi | 2 | 2.06 | pthXo1, pthXo2, avrXa23, avrXa27 |
| III | YN17-2, YN18-6 | Yunnan, Guangdong | 8 | 8.25 | pthXo1, pthXo2, avrXa10, avrXa23, avrXa27 |
| IV | GX15-4, GD3-153 | Guangxi, Guangdong, Yunnan | 18 | 18.56 | pthXo1, pthXo2, avrXa23 |
| V | ZJ1-121, HuN1-136 | Zhejiang, Hunan, Jiangsu | 27 | 27.84 | pthXo1, pthXo2, avrXa23 |
| VI | ZJ3-184 | Zhejiang, Guangdong | 5 | 5.15 | pthXo1, pthXo2, avrXa23, avrXa27 |
| VII | ZJ4-198 | Zhejiang, Jiangsu | 4 | 4.12 | pthXo1, pthXo2, avrXa23, avrXa27 |
| VIII | GD1-105, HN1-150 | Guangdong, Hainan, Zhejiang, Guangxi | 22 | 22.68 | pthXo1, pthXo2, avrXa23 |
| IX | FJ3-192 | Fujian, Guangxi | 4 | 4.12 | pthXo1, avrXa7, avrXa10, avrXa23, avrXa27 |
| X | JS2-103 | Jiangsu, Zhejiang | 3 | 3.09 | pthXo1, pthXo2, avrXa7, avrXa10, avrXa23, avrXa27 |
| Strains | tale genotype | IR24 | JG30 | IRBB3 | IRBB4 | IRBB5 | IRBB7 | IRBB21 | IRBB27 | CBB23 | KΔSWEET | 5024 | 4059 | 4064 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| YN18-3 | I | 2.17± 1.21 b | 11.00± 1.25 a | 0.50± 0.32 b | 0.50± 0.69 b | 0.50± 0.04 b | 0.10± 0.00 b | 0.50± 0.31 b | 0.50± 0.01 b | 0.30± 0.01 b | 1.90± 0.40 b | 0.10± 0.00 b | 0.35± 0.01 b | 0.35± 0.05 b |
| GX15-2 | II | 9.00± 0.69 b | 14.00± 0.82 a | 8.67± 2.75 b | 7.17± 0.05 c | 1.67± 0.02 c | 2.50± 0.01 d | 4.44± 0.45 c | 0.10± 0.00 d | 1.88± 0.03 d | 2.13± 0.45 d | 0.10± 0.00 d | 0.11± 0.02 d | 14.33± 1.25 a |
| YN18-6 | III | 9.83± 0.02 b | 18.83± 0.96 a | 15.67± 0.01 a | 3.50± 0.03 c | 0.40± 0.01 d | 0.48± 0.17 d | 0.50± 0.00 d | 0.32± 0.01 d | 0.10± 0.02 d | 1.40± 0.01 d | 0.20± 0.01 d | 0.60± 0.01 d | 2.42± 0.01 d |
| GD3-153 | IV | 8.17± 0.90 b | 17.00± 2.58 a | 8.67± 2.98 b | 0.50± 0.01 d | 0.10± 0.00 d | 0.10± 0.02 d | 3.38± 0.02 c | 0.50± 0.02 d | 0.10± 0.01 d | 1.08± 1.92 d | 0.10± 0.00 d | 0.10± 0.01 d | 7.00± 1.63 b |
| HuN1-136 | V | 9.50± 0.58 b | 11.33± 2.08 a | 5.17± 0.94 c | 6.75± 1.34 c | 0.10± 0.02 d | 0.10± 0.01 d | 14.83± 1.25 a | 0.20± 0.01 d | 0.10± 0.10 d | 0.75± 0.22 d | 3.50± 0.01 c | 1.17± 0.02 d | 9.92± 1.21 b |
| ZJ3-184 | VI | 6.33± 0.37 b | 14.50± 1.83 a | 7.75± 0.02 b | 0.20± 0.03 c | 0.10± 0.00 c | 2.75± 0.02 c | 0.10± 0.00 c | 0.10± 0.00 c | 0.88± 0.32 c | 0.35± 0.15 c | 0.10± 0.00 c | 1.33± 0.01 c | 2.50± 0.14 c |
| ZJ4-198 | VII | 9.50± 1.21 b | 12.00± 1.07 a | 6.67± 1.70 c | 7.33± 1.61 c | 1.75± 0.32 d | 0.10± 0.01 d | 2.42± 0.03 d | 0.10± 0.00 d | 0.10± 0.01 d | 7.83± 0.21 c | 0.10± 0.02 d | 0.50± 0.01 d | 5.83± 0.25 c |
| HN1-150 | VIII | 10.33± 0.69 b | 17.17± 1.29 a | 14.00± 0.94 a | 6.17± 1.12 c | 0.33± 0.01 d | 0.53± 0.25 d | 3.86± 0.01 d | 8.67± 3.34 c | 1.21± 0.24 d | 1.10± 0.24 d | 4.00± 0.95 d | 4.83± 0.05 c | 8.17± 0.14 c |
| FJ3-192 | IX | 6.67± 1.97 b | 8.17± 1.34 ab | 4.75± 1.15 bc | 4.92± 0.69 bc | 0.10± 0.00 c | 5.08± 0.37 b | 1.00± 0.01 c | 0.10± 0.02 c | 1.42± 1.28 c | 1.50± 0.37 c | 0.83± 0.01 c | 2.67± 0.01 c | 10.33± 0.17 a |
| JS2-103 | X | 4.83± 0.96 c | 12.83± 0.75 a | 7.17± 0.69 b | 1.67± 1.18 d | 0.50± 0.00 d | 1.93± 0.07 d | 2.17± 0.00 d | 0.10± 0.00 d | 0.25± 0.07 d | 1.58± 0.43 d | 0.50± 0.02 d | 0.10± 0.00 d | 4.83± 2.89 c |
表2 携带不同抗性基因的水稻品种中Xoo 10个tale基因型代表菌株的毒力评价
Table 2 Virulence evaluation of 10 tale genotypes of Xoo strains in rice cultivars containing different R genes
| Strains | tale genotype | IR24 | JG30 | IRBB3 | IRBB4 | IRBB5 | IRBB7 | IRBB21 | IRBB27 | CBB23 | KΔSWEET | 5024 | 4059 | 4064 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| YN18-3 | I | 2.17± 1.21 b | 11.00± 1.25 a | 0.50± 0.32 b | 0.50± 0.69 b | 0.50± 0.04 b | 0.10± 0.00 b | 0.50± 0.31 b | 0.50± 0.01 b | 0.30± 0.01 b | 1.90± 0.40 b | 0.10± 0.00 b | 0.35± 0.01 b | 0.35± 0.05 b |
| GX15-2 | II | 9.00± 0.69 b | 14.00± 0.82 a | 8.67± 2.75 b | 7.17± 0.05 c | 1.67± 0.02 c | 2.50± 0.01 d | 4.44± 0.45 c | 0.10± 0.00 d | 1.88± 0.03 d | 2.13± 0.45 d | 0.10± 0.00 d | 0.11± 0.02 d | 14.33± 1.25 a |
| YN18-6 | III | 9.83± 0.02 b | 18.83± 0.96 a | 15.67± 0.01 a | 3.50± 0.03 c | 0.40± 0.01 d | 0.48± 0.17 d | 0.50± 0.00 d | 0.32± 0.01 d | 0.10± 0.02 d | 1.40± 0.01 d | 0.20± 0.01 d | 0.60± 0.01 d | 2.42± 0.01 d |
| GD3-153 | IV | 8.17± 0.90 b | 17.00± 2.58 a | 8.67± 2.98 b | 0.50± 0.01 d | 0.10± 0.00 d | 0.10± 0.02 d | 3.38± 0.02 c | 0.50± 0.02 d | 0.10± 0.01 d | 1.08± 1.92 d | 0.10± 0.00 d | 0.10± 0.01 d | 7.00± 1.63 b |
| HuN1-136 | V | 9.50± 0.58 b | 11.33± 2.08 a | 5.17± 0.94 c | 6.75± 1.34 c | 0.10± 0.02 d | 0.10± 0.01 d | 14.83± 1.25 a | 0.20± 0.01 d | 0.10± 0.10 d | 0.75± 0.22 d | 3.50± 0.01 c | 1.17± 0.02 d | 9.92± 1.21 b |
| ZJ3-184 | VI | 6.33± 0.37 b | 14.50± 1.83 a | 7.75± 0.02 b | 0.20± 0.03 c | 0.10± 0.00 c | 2.75± 0.02 c | 0.10± 0.00 c | 0.10± 0.00 c | 0.88± 0.32 c | 0.35± 0.15 c | 0.10± 0.00 c | 1.33± 0.01 c | 2.50± 0.14 c |
| ZJ4-198 | VII | 9.50± 1.21 b | 12.00± 1.07 a | 6.67± 1.70 c | 7.33± 1.61 c | 1.75± 0.32 d | 0.10± 0.01 d | 2.42± 0.03 d | 0.10± 0.00 d | 0.10± 0.01 d | 7.83± 0.21 c | 0.10± 0.02 d | 0.50± 0.01 d | 5.83± 0.25 c |
| HN1-150 | VIII | 10.33± 0.69 b | 17.17± 1.29 a | 14.00± 0.94 a | 6.17± 1.12 c | 0.33± 0.01 d | 0.53± 0.25 d | 3.86± 0.01 d | 8.67± 3.34 c | 1.21± 0.24 d | 1.10± 0.24 d | 4.00± 0.95 d | 4.83± 0.05 c | 8.17± 0.14 c |
| FJ3-192 | IX | 6.67± 1.97 b | 8.17± 1.34 ab | 4.75± 1.15 bc | 4.92± 0.69 bc | 0.10± 0.00 c | 5.08± 0.37 b | 1.00± 0.01 c | 0.10± 0.02 c | 1.42± 1.28 c | 1.50± 0.37 c | 0.83± 0.01 c | 2.67± 0.01 c | 10.33± 0.17 a |
| JS2-103 | X | 4.83± 0.96 c | 12.83± 0.75 a | 7.17± 0.69 b | 1.67± 1.18 d | 0.50± 0.00 d | 1.93± 0.07 d | 2.17± 0.00 d | 0.10± 0.00 d | 0.25± 0.07 d | 1.58± 0.43 d | 0.50± 0.02 d | 0.10± 0.00 d | 4.83± 2.89 c |
| [1] | 张华, 姜英华, 胡白石, 刘凤权, 许志刚 (2008). 利用PCR技术专化性检测水稻细菌性条斑病菌. 植物病理学报 38, 1-5. |
| [2] |
Antony G, Zhou JH, Huang S, Li T, Liu B, White F, Yang B (2010). Rice xa13 recessive resistance to bacterial blight is defeated by induction of the disease susceptibility gene Os-11N3. Plant Cell 22, 3864-3876.
DOI URL |
| [3] | Boch J, Bonas U, Lahaye T (2014). TAL effectors-pathogen strategies and plant resistance engineering. New Phy- tol 204, 823-832. |
| [4] |
Breia R, Conde A, Badim H, Fortes AM, Gerós H, Granell A (2021). Plant SWEETs: from sugar transport to plant-pathogen interaction and more unexpected physiological roles. Plant Physiol 186, 836-852.
DOI PMID |
| [5] |
Doyle EL, Stoddard BL, Voytas DF, Bogdanove AJ (2013). TAL effectors: highly adaptable phytobacterial virulence factors and readily engineered DNA-targeting proteins. Trends Cell Biol 23, 390-398.
DOI PMID |
| [6] |
Ji CH, Ji ZY, Liu B, Cheng H, Liu H, Liu SZ, Yang B, Chen GY (2020). Xa1 Allelic R genes activate rice blight resistance suppressed by interfering TAL effectors. Plant Commun 1, 100087.
DOI URL |
| [7] |
Ji ZY, Guo W, Chen XF, Wang CL, Zhao KJ (2022). Plant executor genes. Int J Mol Sci 23, 1524.
DOI URL |
| [8] |
Ji ZY, Ji CH, Liu B, Zou LF, Chen GY, Yang B (2016). Interfering TAL effectors of Xanthomonas oryzae neutralize R-gene-mediated plant disease resistance. Nat Commun 7, 13435.
DOI |
| [9] |
Ji ZY, Wang CL, Zhao KJ (2018). Rice routes of countering Xanthomonas oryzae. Int J Mol Sci 19, 3008.
DOI URL |
| [10] |
Ji ZY, Zakria M, Zou LF, Xiong L, Li Z, Ji GH, Chen GY (2014). Genetic diversity of transcriptional activator-like effector genes in Chinese isolates of Xanthomonas oryzae pv. oryzicola. Phytopathology 104, 672-682.
DOI URL |
| [11] |
Niño-Liu DO, Ronald PC, Bogdanove AJ (2006). Xanthomonas oryzae pathovars: model pathogens of a model crop. Mol Plant Pathol 7, 303-324.
DOI PMID |
| [12] |
Nowack MK, Holmes DR, Lahaye T (2022). TALE-induced cell death executors: an origin outside immunity? Trends Plant Sci 27, 536-548.
DOI URL |
| [13] |
Oliva R, Ji CH, Atienza-Grande G, Huguet-Tapia JC, Perez-Quintero A, Li T, Eom JS, Li CH, Nguyen H, Liu B, Auguy F, Sciallano C, Luu VT, Dossa GS, Cunnac S, Schmidt SM, Slamet-Loedin IH, Vera Cruz C, Szurek B, Frommer WB, White FF, Yang B (2019). Broad- spectrum resistance to bacterial blight in rice using genome editing. Nat Biotechnol 37, 1344-1350.
DOI |
| [14] |
Perez-Quintero AL, Szurek B (2019). A decade decoded: spies and hackers in the history of TAL effectors research. Annu Rev Phytopathol 57, 459-481.
DOI PMID |
| [15] | Sambrook J, Fritsch EF, Maniatis T (1989). Molecular Cloning: A Laboratory Manual, 2nd edn. Cold Spring Harbor: Cold Spring Harbor Laboratory. |
| [16] | Wang CL, Qin TF, Yu HM, Zhang XP, Che JY, Gao Y, Zheng CK, Yang B, Zhao KJ (2014). The broad bacterial blight resistance of rice line CBB23 is triggered by a novel transcription activator-like (TAL) effector of Xanthomonas oryzae pv. oryzae. Mol Plant Pathol 15, 333-341. |
| [17] |
White FF, Yang B (2009). Host and pathogen factors controlling the rice-Xanthomonas oryzae interaction. Plant Physiol 150, 1677-1686.
DOI URL |
| [18] |
Xu ZY, Xu XM, Gong Q, Li ZY, Li Y, Wang S, Yang YY, Ma WX, Liu LY, Zhu B, Zou LF, Chen GY (2019). Engineering broad-spectrum bacterial blight resistance by simultaneously disrupting variable TALE-binding elements of multiple susceptibility genes in rice. Mol Plant 12, 1434-1446.
DOI PMID |
| [19] |
Xu ZY, Zou LF, Ma WX, Cai LL, Yang YY, Chen GY (2017). Action modes of transcription activator-like effectors (TALEs) of Xanthomonas in plants. J Integr Agric 16, 2736-2745.
DOI URL |
| [20] |
Yang B, Sugio A, White FF (2006). Os8N3 is a host disease-susceptibility gene for bacterial blight of rice. Proc Natl Acad Sci USA 103, 10503-10508.
DOI URL |
| [21] |
Zhang BM, Zhang HT, Li F, Ouyang YD, Yuan M, Li XH, Xiao JH, Wang SP (2020). Multiple alleles encoding atypical NLRs with unique central tandem repeats in rice confer resistance to Xanthomonas oryzae pv. oryzae. Plant Commun 1, 100088.
DOI URL |
| [22] |
Zhou JH, Peng Z, Long JY, Sosso D, Liu B, Eom JS, Huang S, Liu SZ, Vera Cruz C, Frommer WB, White FF, Yang B (2015). Gene targeting by the TAL effector PthXo2 reveals cryptic resistance gene for bacterial blight of rice. Plant J 82, 632-643.
DOI URL |
| [23] |
Zhou JM, Zhang YL (2020). Plant immunity: danger perception and signaling. Cell 181, 978-989.
DOI URL |
| [1] | 叶灿, 姚林波, 金莹, 高蓉, 谭琪, 李旭映, 张艳军, 陈析丰, 马伯军, 章薇, 张可伟. 水稻水杨酸代谢突变体高通量筛选方法的建立与应用[J]. 植物学报, 2025, 60(4): 1-0. |
| [2] | 赵凌, 管菊, 梁文化, 张勇, 路凯, 赵春芳, 李余生, 张亚东. 基于高密度Bin图谱的水稻苗期耐热性QTL定位[J]. 植物学报, 2025, 60(3): 342-353. |
| [3] | 李新宇, 谷月, 徐非非, 包劲松. 水稻胚乳淀粉合成相关蛋白的翻译后修饰研究进展[J]. 植物学报, 2025, 60(2): 256-270. |
| [4] | 李建国, 张怡, 张文君. 水稻根系铁膜形成及对磷吸收的影响[J]. 植物学报, 2025, 60(1): 132-143. |
| [5] | 姚瑞枫, 谢道昕. 水稻独脚金内酯信号感知的激活和终止[J]. 植物学报, 2024, 59(6): 873-877. |
| [6] | 连锦瑾, 唐璐瑶, 张伊诺, 郑佳兴, 朱超宇, 叶语涵, 王跃星, 商文楠, 傅正浩, 徐昕璇, 吴日成, 路梅, 王长春, 饶玉春. 水稻抗氧化性状遗传位点挖掘及候选基因分析[J]. 植物学报, 2024, 59(5): 738-751. |
| [7] | 黄佳慧, 杨惠敏, 陈欣雨, 朱超宇, 江亚楠, 胡程翔, 连锦瑾, 芦涛, 路梅, 张维林, 饶玉春. 水稻突变体pe-1对弱光胁迫的响应机制[J]. 植物学报, 2024, 59(4): 574-584. |
| [8] | 周俭民. 收放自如的明星战车[J]. 植物学报, 2024, 59(3): 343-346. |
| [9] | 朱超宇, 胡程翔, 朱哲楠, 张芷宁, 汪理海, 陈钧, 李三峰, 连锦瑾, 唐璐瑶, 钟芊芊, 殷文晶, 王跃星, 饶玉春. 水稻穗部性状QTL定位及候选基因分析[J]. 植物学报, 2024, 59(2): 217-230. |
| [10] | 夏婧, 饶玉春, 曹丹芸, 王逸, 柳林昕, 徐雅婷, 牟望舒, 薛大伟. 水稻中乙烯生物合成关键酶OsACS和OsACO调控机制研究进展[J]. 植物学报, 2024, 59(2): 291-301. |
| [11] | 朱宝, 赵江哲, 张可伟, 黄鹏. 水稻细胞分裂素氧化酶9参与调控水稻叶夹角发育[J]. 植物学报, 2024, 59(1): 10-21. |
| [12] | 方妍力, 田传玉, 苏如意, 刘亚培, 王春连, 陈析丰, 郭威, 纪志远. 水稻抗细菌性条斑病基因挖掘与初定位[J]. 植物学报, 2024, 59(1): 1-9. |
| [13] | 贾绮玮, 钟芊芊, 顾育嘉, 陆天麒, 李玮, 杨帅, 朱超宇, 胡程翔, 李三峰, 王跃星, 饶玉春. 水稻茎秆细胞壁相关组分含量QTL定位及候选基因分析[J]. 植物学报, 2023, 58(6): 882-892. |
| [14] | 戴若惠, 钱心妤, 孙静蕾, 芦涛, 贾绮玮, 陆天麒, 路梅, 饶玉春. 水稻叶色调控机制及相关基因研究进展[J]. 植物学报, 2023, 58(5): 799-812. |
| [15] | 严语萍, 俞晓琦, 任德勇, 钱前. 水稻穗粒数遗传机制与育种利用[J]. 植物学报, 2023, 58(3): 359-372. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||