

植物学报 ›› 2022, Vol. 57 ›› Issue (3): 276-287.DOI: 10.11983/CBB22018 cstr: 32102.14.CBB22018
杨凯如1, 贾绮玮1, 金佳怡1, 叶涵斐1, 王盛1, 陈芊羽1, 管易安1, 潘晨阳1, 辛德东1, 方媛1,*( ), 王跃星2,*(
), 王跃星2,*( ), 饶玉春1,*(
), 饶玉春1,*( )
)
收稿日期:2022-01-19
接受日期:2022-03-18
出版日期:2022-05-01
发布日期:2022-05-18
通讯作者:
方媛,王跃星,饶玉春
作者简介:ryc@zjnu.cn基金资助:
Kairu Yang1, Qiwei Jia1, Jiayi Jin1, Hanfei Ye1, Sheng Wang1, Qianyu Chen1, Yian Guan1, Chenyang Pan1, Dedong Xin1, Yuan Fang1,*( ), Yuexing Wang2,*(
), Yuexing Wang2,*( ), Yuchun Rao1,*(
), Yuchun Rao1,*( )
)
Received:2022-01-19
Accepted:2022-03-18
Online:2022-05-01
Published:2022-05-18
Contact:
Yuan Fang,Yuexing Wang,Yuchun Rao
摘要: 叶色突变体往往伴随着叶绿素含量变化及叶绿体结构异常, 是研究叶绿体发育与光合作用相关基因功能的重要材料。该研究通过甲基磺酸乙酯(EMS)诱变籼稻(Oryza sativa subsp. indica)品种华占(HZ)获得黄绿叶突变体, 将其命名为ygl18 (yellow-green leaf 18)。与野生型相比, 黄绿叶突变体ygl18自三叶期起叶片开始变黄且程度不断加深, 同时伴随着光合速率与叶绿素含量下降, 且结实率、千粒重及有效穗数均显著降低。透射电镜观察结果显示, ygl18的叶绿体结构紊乱, 基质片层疏松, 发育受到抑制, 与叶片出现黄绿色表型一致。遗传分析表明, ygl18突变性状受1对隐性等位核基因控制, 这对等位基因位于水稻第3号染色体长臂标记InDel2和InDel3之间115.2 kb范围内。进一步研究发现该突变体表型是编码铁氧还蛋白FdC2的基因LOC_Os03g48040的5'UTR发生突变所致。通过CRISPR转基因实验验证了该基因对表型的控制作用。研究结果揭示了叶色调控网络的遗传基础, 可为今后选育高光效水稻品种提供新线索。
杨凯如, 贾绮玮, 金佳怡, 叶涵斐, 王盛, 陈芊羽, 管易安, 潘晨阳, 辛德东, 方媛, 王跃星, 饶玉春. 水稻黄绿叶调控基因YGL18的克隆与功能解析. 植物学报, 2022, 57(3): 276-287.
Kairu Yang, Qiwei Jia, Jiayi Jin, Hanfei Ye, Sheng Wang, Qianyu Chen, Yian Guan, Chenyang Pan, Dedong Xin, Yuan Fang, Yuexing Wang, Yuchun Rao. Cloning and Functional Analysis of Rice Yellow Green Leaf Regulatory Gene YGL18. Chinese Bulletin of Botany, 2022, 57(3): 276-287.
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) |
|---|---|---|
| B3-14 | AGGATTTGGGCTGTCAATGC | AGCCTACCACAAGACATATAATCG |
| B3-15 | TCCAGTCCACTAAAAGTTTTC | ATTATGCTATAGGCTTTGAGC |
| M1 | TGGAAATGGGTGGAATACCT | CCTCTGTGAGGACCATCTCC |
| M2 | GAATTATTTTTCCTTGGTCATTGTG | GTACCAACGAGGGTCCAAAG |
| M3 | GGAGGCAGGCTACACCAGTA | GCACAATTCTTAAAGAGATGAACC |
| M4 | TTTGATTGGGCTCAAAGCAC | GCCAGGATTTGTTGGATTAGG |
| M5 | AAAAACTCCCAGCGGATAGA | GCTAGAACTGGCAAAATGCTG |
| Indel1 | TTAGGCAAATGAAACCAAGG | CCCAACCACCTAAGTAAACGA |
| Indel2 | TCCTTTGATGACTGGTCGGC | ATGGGAAGACGGGGGAGTAA |
| Indel3 | GTTTGGGAGAGACATGGGGG | GTTTGGCCCATTCCCTGAAG |
| Indel4 | ATGATTTGGAGCCAGCAGTT | GGAAGTGGAATTGCAGATGG |
表1 精细定位所用引物序列
Table 1 Primer sequences for fine mapping
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) |
|---|---|---|
| B3-14 | AGGATTTGGGCTGTCAATGC | AGCCTACCACAAGACATATAATCG |
| B3-15 | TCCAGTCCACTAAAAGTTTTC | ATTATGCTATAGGCTTTGAGC |
| M1 | TGGAAATGGGTGGAATACCT | CCTCTGTGAGGACCATCTCC |
| M2 | GAATTATTTTTCCTTGGTCATTGTG | GTACCAACGAGGGTCCAAAG |
| M3 | GGAGGCAGGCTACACCAGTA | GCACAATTCTTAAAGAGATGAACC |
| M4 | TTTGATTGGGCTCAAAGCAC | GCCAGGATTTGTTGGATTAGG |
| M5 | AAAAACTCCCAGCGGATAGA | GCTAGAACTGGCAAAATGCTG |
| Indel1 | TTAGGCAAATGAAACCAAGG | CCCAACCACCTAAGTAAACGA |
| Indel2 | TCCTTTGATGACTGGTCGGC | ATGGGAAGACGGGGGAGTAA |
| Indel3 | GTTTGGGAGAGACATGGGGG | GTTTGGCCCATTCCCTGAAG |
| Indel4 | ATGATTTGGAGCCAGCAGTT | GGAAGTGGAATTGCAGATGG |
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) |
|---|---|---|
| CAO1-qrt | GCTGCTCTACCGGATGTCTC | ACAACCGATACCGGATACCA |
| DVR-qrt | TTCACCTACAGCATCGTCCG | AACAGCATCTCCCCTTGCTC |
| YGL1-qrt | TGTTGGTGGGTCCTTGCTTT | ACTGAAGCCCCAGAGCTCTA |
| PORA-qrt | CATGCTCGACGACCTCAAGA | TCCTGCATCGTCAGCATGTT |
| PORB-qrt | CATCATCGTCGGCTCCATCA | TCCTGCATCGTCAGCATGTT |
| ChlH-qrt | GGGAACTTGGCGTTTCATTA | TCAAATGTCTTGCGTTGCTC |
| ChlD-qrt | TCCTTTAGCTCACGGCCTTA | GTCCAACATCACCGCTCTTT |
| NOL-qrt | TCACAAGCCTTACGACCCAC | GCTCCCTCTCCATCATCTGC |
| NYC1-qrt | TAGTTGGCTCGGGGGAGTAA | AAGGACTGATTCAGGGCTGC |
| OsFdC2-qrt | AGCTGTGCGGTTCGGATAAA | AACAGGTCCTCGTGCGAAAT |
| rpoA-qrt | CCATTCCCACAAGCAAAAAT | TCTTACCGCCTTCCGTAGAA |
| rpoB-qrt | TGGTACATATCCCTTATCTCAA | CTCCAGGACCCAAACAACTC |
| rbcL-qrt | CTTGGCAGCATTCCGAGTAA | ACAACGGGCTCGATGTGATA |
| rbcS-qrt | GCTTGGAGTTCAGCAAGGTC | AACGAAGGCATCAGGGTATG |
| PsaA-qrt | TTAGAAATCCGCCAATCCA | TGCTAGGCTCTACAACCATT |
| PsaD-qrt | CCGCTCCAAGTACAAGATCA | AAGAGCAGCCTGACAGATGA |
| PsbA-qrt | ACCCTCATTAGCAGATTCGT | GATTGTATTCCAGGCAGAGC |
| PsbD-qrt | AAGACAGATTCCGAGGGTGG | TGATTCGCTAGGGATTAAAGAG |
表2 qRT-PCR所用引物序列
Table 2 Primer sequences for qRT-PCR
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) |
|---|---|---|
| CAO1-qrt | GCTGCTCTACCGGATGTCTC | ACAACCGATACCGGATACCA |
| DVR-qrt | TTCACCTACAGCATCGTCCG | AACAGCATCTCCCCTTGCTC |
| YGL1-qrt | TGTTGGTGGGTCCTTGCTTT | ACTGAAGCCCCAGAGCTCTA |
| PORA-qrt | CATGCTCGACGACCTCAAGA | TCCTGCATCGTCAGCATGTT |
| PORB-qrt | CATCATCGTCGGCTCCATCA | TCCTGCATCGTCAGCATGTT |
| ChlH-qrt | GGGAACTTGGCGTTTCATTA | TCAAATGTCTTGCGTTGCTC |
| ChlD-qrt | TCCTTTAGCTCACGGCCTTA | GTCCAACATCACCGCTCTTT |
| NOL-qrt | TCACAAGCCTTACGACCCAC | GCTCCCTCTCCATCATCTGC |
| NYC1-qrt | TAGTTGGCTCGGGGGAGTAA | AAGGACTGATTCAGGGCTGC |
| OsFdC2-qrt | AGCTGTGCGGTTCGGATAAA | AACAGGTCCTCGTGCGAAAT |
| rpoA-qrt | CCATTCCCACAAGCAAAAAT | TCTTACCGCCTTCCGTAGAA |
| rpoB-qrt | TGGTACATATCCCTTATCTCAA | CTCCAGGACCCAAACAACTC |
| rbcL-qrt | CTTGGCAGCATTCCGAGTAA | ACAACGGGCTCGATGTGATA |
| rbcS-qrt | GCTTGGAGTTCAGCAAGGTC | AACGAAGGCATCAGGGTATG |
| PsaA-qrt | TTAGAAATCCGCCAATCCA | TGCTAGGCTCTACAACCATT |
| PsaD-qrt | CCGCTCCAAGTACAAGATCA | AAGAGCAGCCTGACAGATGA |
| PsbA-qrt | ACCCTCATTAGCAGATTCGT | GATTGTATTCCAGGCAGAGC |
| PsbD-qrt | AAGACAGATTCCGAGGGTGG | TGATTCGCTAGGGATTAAAGAG |
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) |
|---|---|---|
| CRISPR/Cas9 | CTCCCTCTCTCGATCGTCAG | GCTACTGCTGCTTGAGACGG |
表3 CRISPR/Cas9载体所用引物
Table 3 Primer sequences for CRISPR/Cas9
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) |
|---|---|---|
| CRISPR/Cas9 | CTCCCTCTCTCGATCGTCAG | GCTACTGCTGCTTGAGACGG |

图1 水稻野生型HZ与突变体ygl18的表型 (A) 分蘖期植株(bar=6 cm); (B) 成熟期植株(bar=10 cm)
Figure 1 Phenotypic of wild-type HZ and mutant ygl18 of rice (A) Phenotype of plant at tillering stage (bar=6 cm); (B) Phenotype of plant at maturity stage (bar=10 cm)
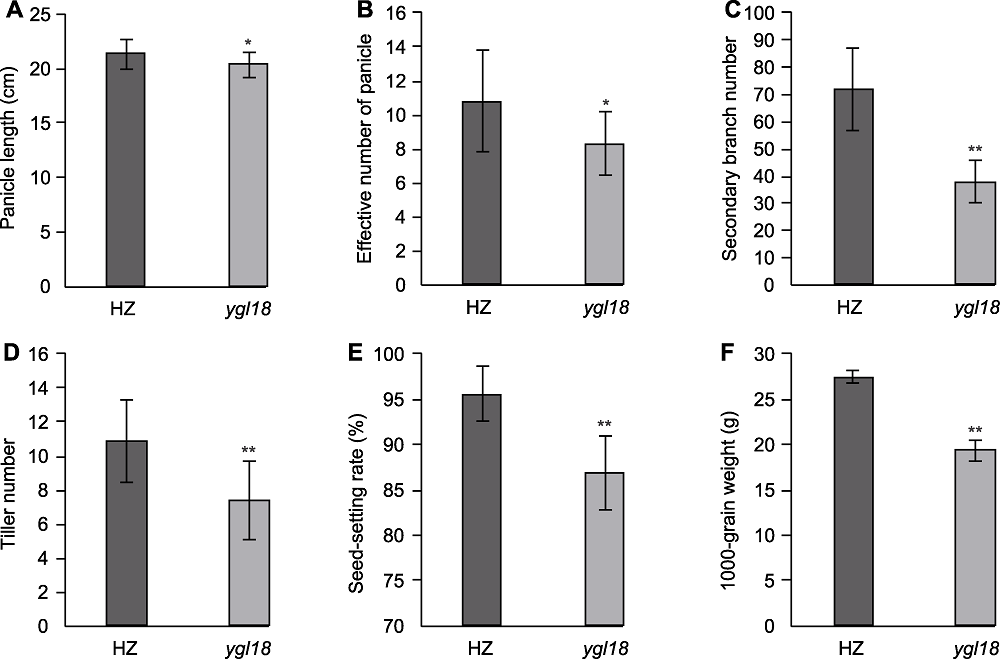
图2 水稻野生型HZ与突变体ygl18的农艺性状 (A) 穗长; (B) 有效穗数; (C) 二次枝梗数; (D) 分蘖数; (E) 结实率; (F) 千粒重。*和**分别表示在0.05和0.01水平差异显著。
Figure 2 Agronomic characters of wild-type HZ and mutant ygl18 of rice (A) Panicle length; (B) Effective number of panicle; (C) Secondary branch number; (D) Tiller number; (E) Seed-setting rate; (F) 1000-grain weight. * and ** indicate significant differences at 0.05 and 0.01 levels, respectively.
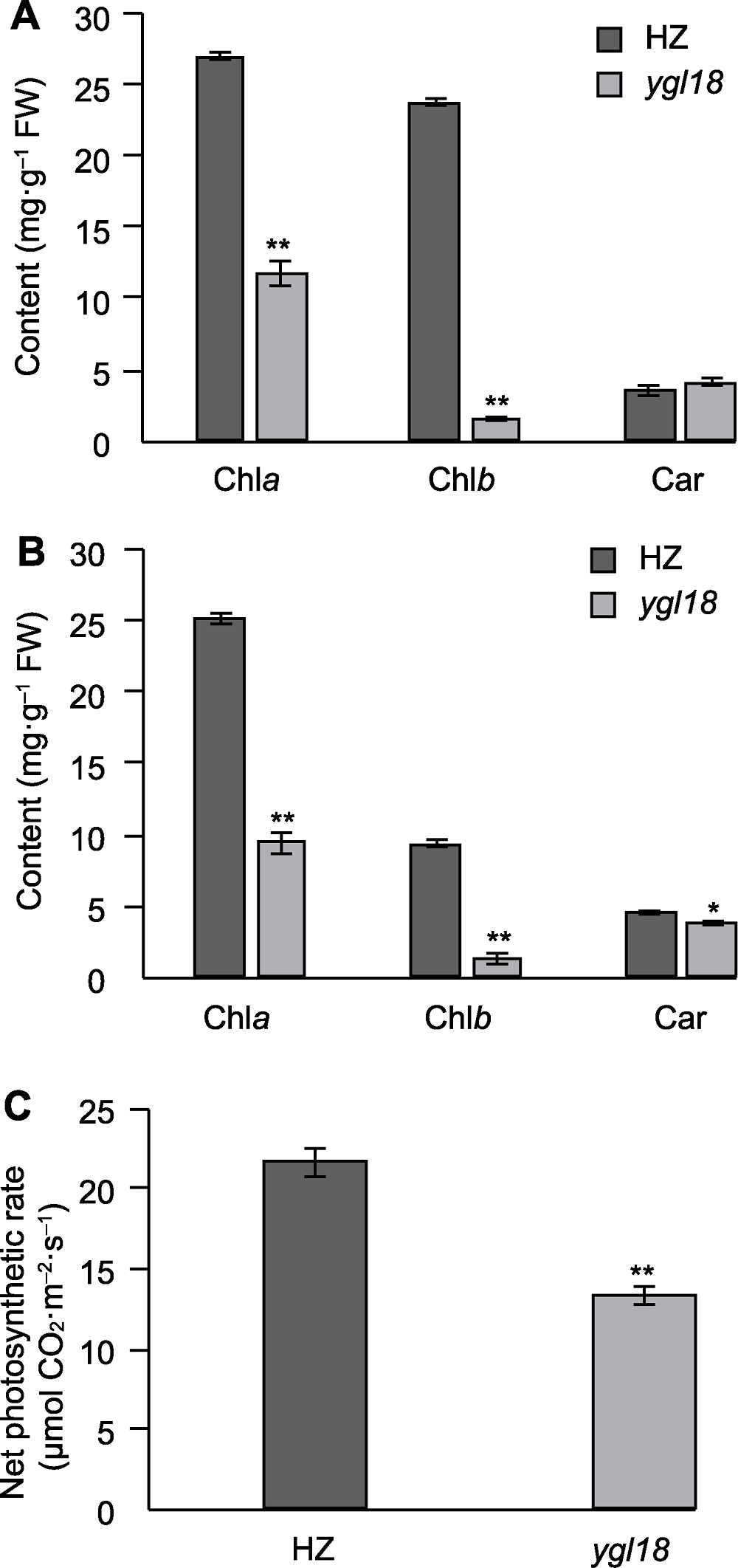
图3 水稻野生型HZ和突变体ygl18叶片光合色素含量和净光合速率 (A) 分蘖期叶片光合色素含量; (B) 抽穗期叶片光合色素含量; (C) 叶片净光合速率。*和**分别表示在0.05和0.01水平差异显著。
Figure 3 Photosynthetic pigment contents and net photosynthetic rate in leaves of wild-type HZ and mutant ygl18 of rice (A) Photosynthetic pigment content of leaves at tillering stage; (B) Photosynthetic pigment content of leaves at heading stage; (C) Net photosynthetic rate of leaves. * and ** indicate significant differences at 0.05 and 0.01 levels, respectively.
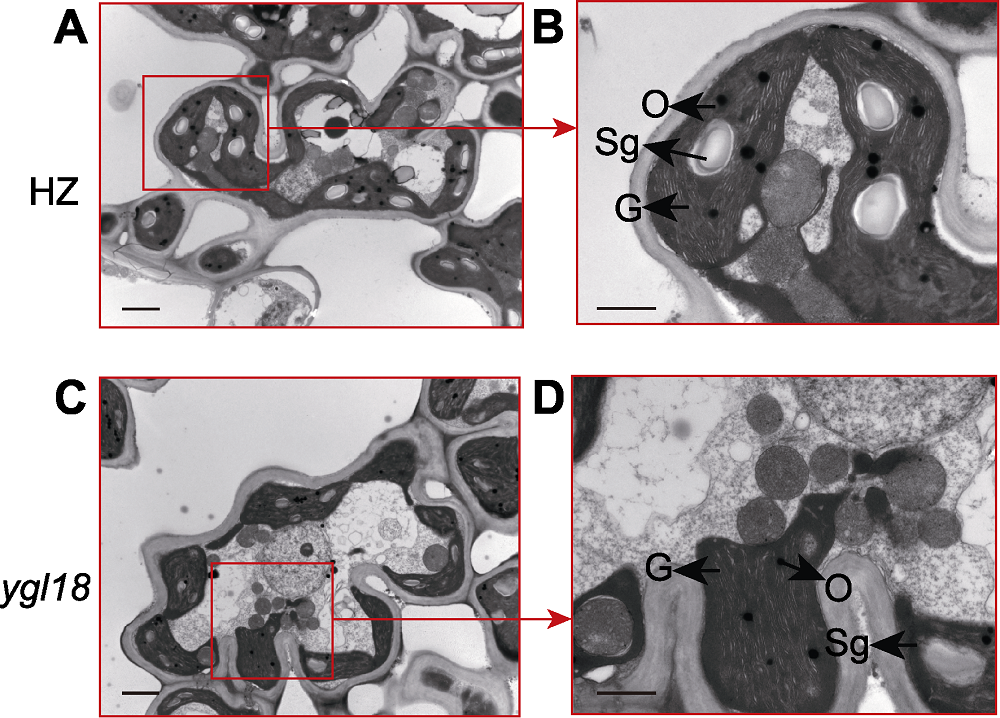
图4 水稻野生型HZ和突变体ygl18叶肉细胞超微结构观察 (A) HZ叶肉细胞(bar=2 μm); (B) HZ叶肉细胞(bar=1 μm); (C) ygl18叶肉细胞(bar=2 μm); (D) ygl18叶肉细胞(bar=1 μm)。G: 类囊体片层; Sg: 淀粉粒; O: 嗜锇粒
Figure 4 Ultrastructural observation of mesophyll cells in wild-type HZ and mutant ygl18 of rice (A) HZ mesophyll cells (bar=2 μm); (B) HZ mesophyll cells (bar=1 μm); (C) ygl18 mesophyll cells (bar=2 μm); (D) ygl18 mesophyll cells (bar=1 μm). G: Grana lamella; Sg: Starch granule; O: Ophilic granule
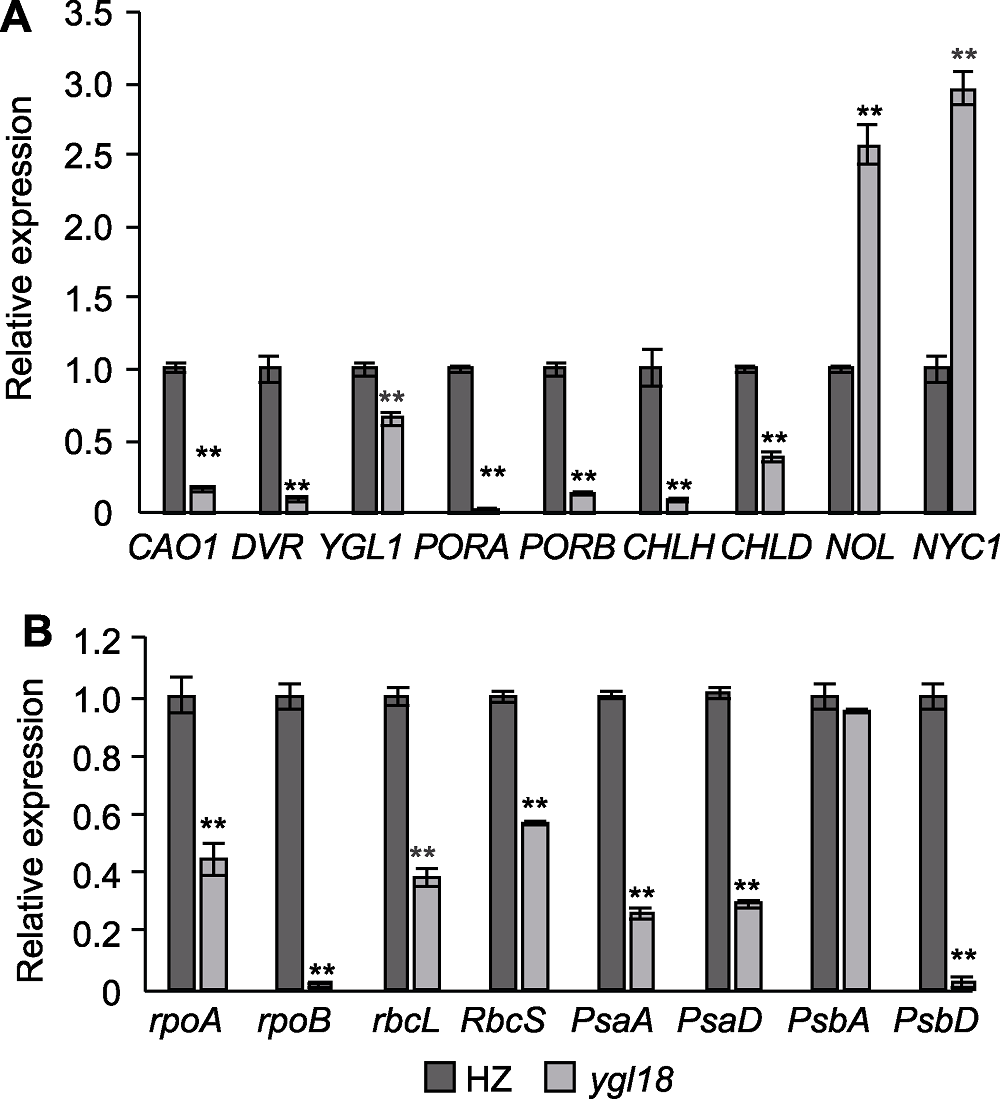
图5 水稻野生型HZ和突变体ygl18分蘖期叶绿素合成、叶绿体发育及光合作用相关基因的表达量 (A) 叶绿素合成相关基因的表达量; (B) 叶绿体发育及光合作用相关基因的表达量
Figure 5 Expression levels of chlorophyll synthesis, chloroplast development and photosynthesis related genes in wild-type HZ and mutant ygl18 of rice at tillering stage (A) Expression of genes related to chlorophyll synthesis; (B) Expression of genes related to chloroplast development and photosynthesis
| Cross | No. of F2 individuals | χ20.05<3.841 | ||
|---|---|---|---|---|
| Normal | yellow-green leaf | Total | ||
| ygl18 × NIP | 836 | 331 | 1167 | 0.833 |
表4 水稻突变体F2代群体的遗传分析
Table 4 Genetic analysis of F2 population of rice mutant
| Cross | No. of F2 individuals | χ20.05<3.841 | ||
|---|---|---|---|---|
| Normal | yellow-green leaf | Total | ||
| ygl18 × NIP | 836 | 331 | 1167 | 0.833 |
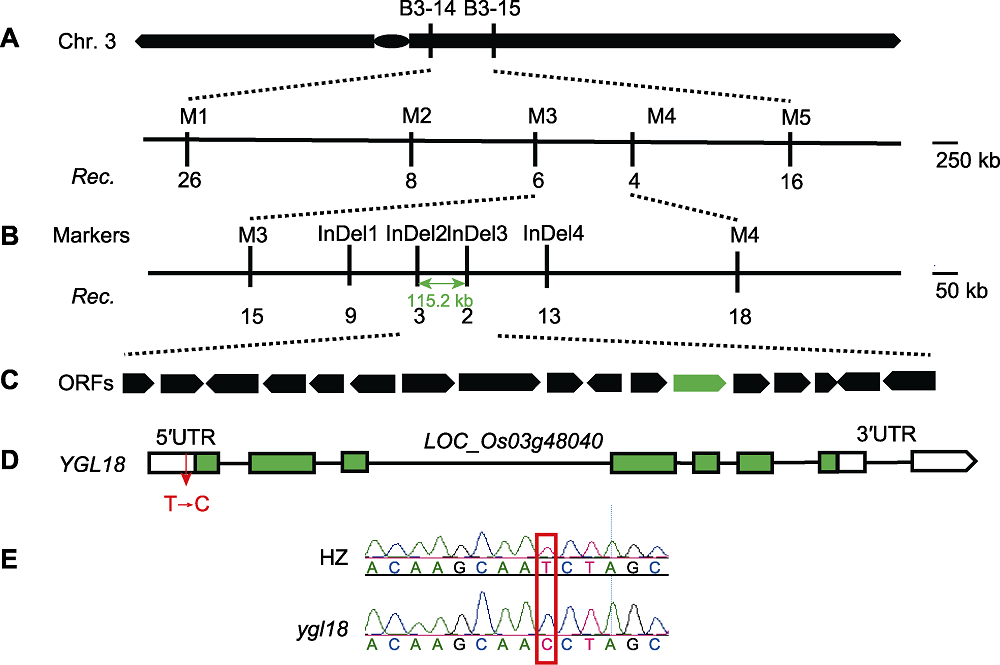
图6 水稻YGL18的精细定位 (A) YGL18定位于水稻3号染色体长臂B3-14与B3-15之间; (B) 使用148个F2代突变个体将其缩小至M3与M4之间; (C) 使用504个F2代突变个体将其限定在InDel2和InDel3之间约115.2 kb的区域中, 共15个开放阅读框(ORFs), 其中绿色部分表示突变基因所在ORF; (D) YGL18基因结构模型(绿色方框表示外显子, 白色方框表示UTRs, 黑线表示内含子); (E) YGL18在野生型和突变体间的测序结果峰图
Figure 6 Fine mapping of YGL18 in rice (A) YGL18 was located between B3-14 and B3-15 on long arm of rice chromosome 3; (B) Then it was narrowed to a region between M3 and M4 using 148 F2 mutants; (C) Then it was limited to an area of about 115.2 kb between InDel2 and InDel3 with a total of 15 open reading frames (ORFs), using 504 F2 mutants; (D) YGL18 gene structure modeling (green boxes indicate exons, white boxes indicate UTRs, and black lines between boxes indicate introns); (E) Peak sequencing result of YGL18 gene between wild type and mutant
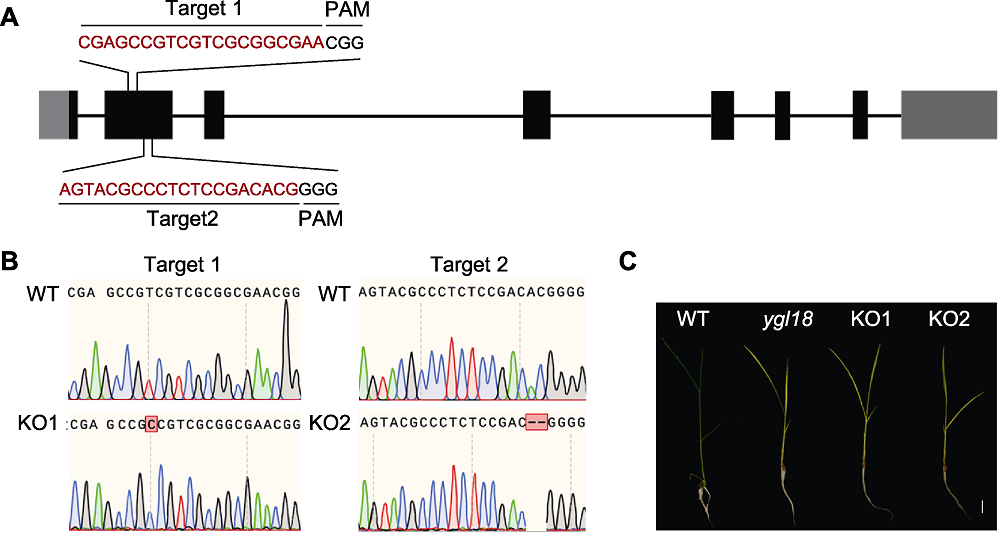
图7 水稻YGL18基因的CRISPR/Cas9敲除验证 (A) 2个分离的敲除靶点位置(黑色方框表示外显子, 灰色方框表示UTRs, 黑线表示内含子, Target 1和Target 2为2个分离的靶点); (B) 基因敲除株的测序验证; (C) 野生型(WT)、突变体ygl18、敲除1及敲除2株系表型(bar=2 cm)。PAM: 原间隔序列邻近基序
Figure 7 CRISPR/Cas9 knockout verification of YGL18 in rice (A) The position of two separate knockout targets (black boxes represent exons, gray boxes represent UTRs, black lines represent introns, and Target 1 and Target 2 are two separate targets); (B) Sequencing verification of gene knockout strains; (C) Phenotype of wild type (WT), mutant ygl18, KO1 and KO2 lines (bar=2 cm). PAM: Protospacer adjacent motif
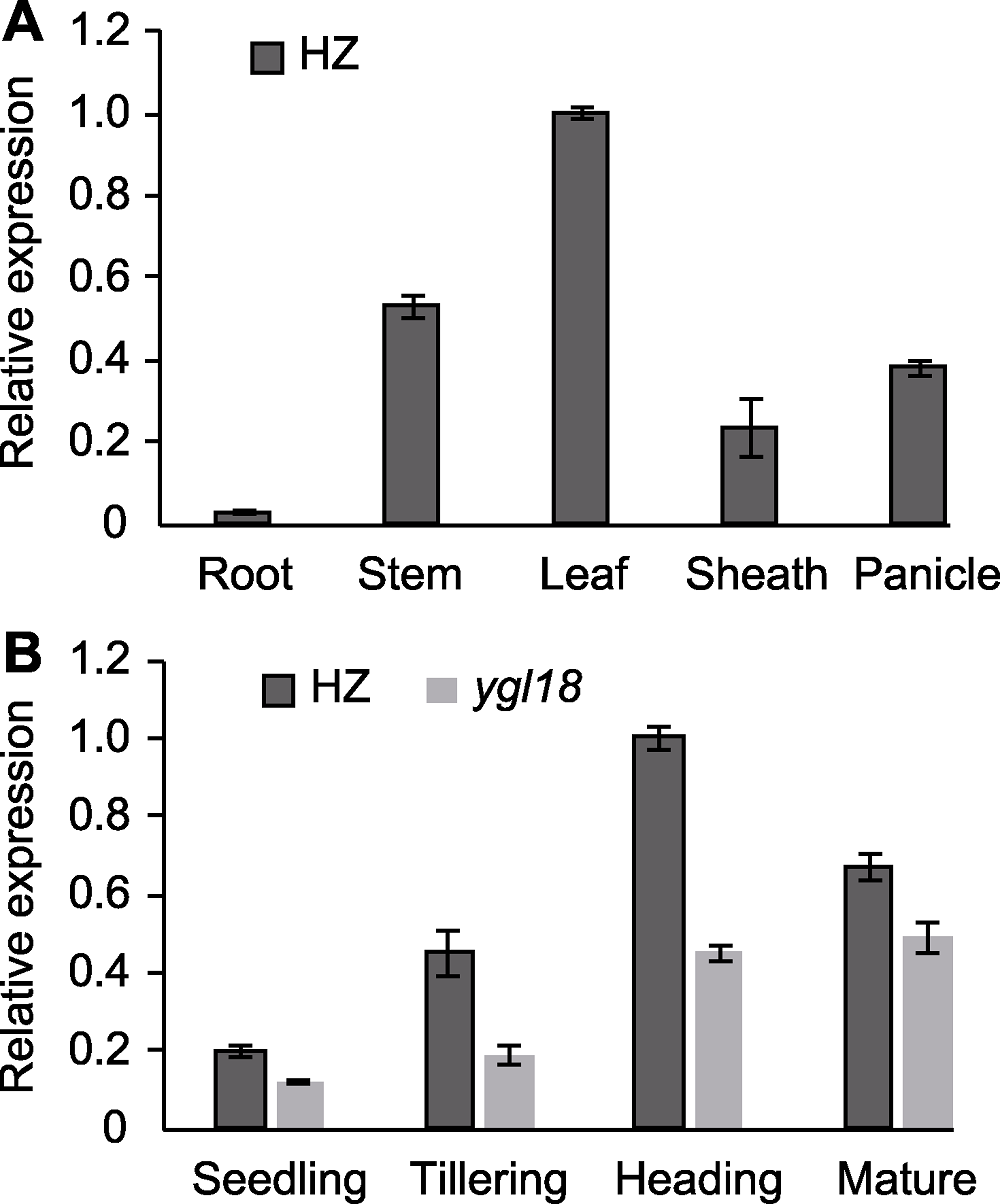
图8 水稻野生型HZ和突变体ygl18中YGL18基因表达模式 (A) YGL18的组织特异性表达; (B) YGL18在不同发育时期的表达水平
Figure 8 Expression pattern of gene YGL18 in wild-type HZ and mutant ygl18 of rice (A) Tissue specific expression of YGL18; (B) The expression level of YGL18 in different development stage
| [1] | 陈昆松, 李方, 徐昌杰, 张上隆, 傅承新 (2004). 改良CTAB法用于多年生植物组织基因组DNA的大量提取. 遗传 26, 529-531. |
| [2] | 杜芳芳, 马骏杰, 杨泽伟, 赵雪莲, 刘东宇, 杨秀芹 (2021). 5'UTR在基因表达调控中的研究进展. 中国畜牧杂志 57(8), 60-67. |
| [3] | 赫磊 (2020). 铁氧还蛋白调控水稻光合电子传递的分子机理研究. 博士论文. 北京: 中国农业科学院. pp. 1-101. |
| [4] |
林添资, 孙立亭, 景德道, 钱华飞, 余波, 曾生元, 李闯, 龚红兵 (2018). 一个水稻黄绿叶突变体ygl14(t)的鉴定及基因定位. 核农学报 32, 216-226.
DOI |
| [5] | 任永梅, 王嘉宇, 王琬璐, 王棋, 姜鑫, 刘振宇, 朱琳 (2019). 水稻白条纹叶突变体wsl1的生理特性分析及基因定位. 沈阳农业大学学报 50, 87-92. |
| [6] | 韦敏益, 吴子帅, 刘立龙, 李允振, 农春晓, 覃宝祥, 李容柏 (2016). 一个水稻长芒基因AWN-2的定位与克隆. 基因组学与应用生物学 35, 949-956. |
| [7] |
徐娜, 徐江民, 蒋玲欢, 饶玉春 (2017). 水稻叶片早衰成因及分子机理研究进展. 植物学报 52, 102-112.
DOI |
| [8] | 许子怡, 程行, 沈奇, 赵亚男, 汤佳玉, 刘喜 (2021). 水稻黄绿叶突变体ygl3的鉴定与基因功能分析. 中国农业科学 54, 3149-3157. |
| [9] | 杨颜榕, 黄纤纤, 赵亚男, 汤佳玉, 刘喜 (2020). 水稻叶色基因克隆与分子机制研究进展. 植物遗传资源学报 21, 794- 803. |
| [10] | 张芳燕, 罗翔, 董娜, 张鹏 (2011). 5'UTR与基因表达的关系. 科技经济市场 (3), 13-16. |
| [11] | 张力科, 高用明 (2009). 水稻叶色突变体及其基因定位和克隆的研究进展. 作物杂志 (2), 12-16. |
| [12] |
周纯, 焦然, 胡萍, 林晗, 胡娟, 徐娜, 吴先美, 饶玉春, 王跃星 (2019). 水稻早衰突变体LS-es1的基因定位及候选基因分析. 植物学报 54, 606-619.
DOI |
| [13] |
周亭亭, 饶玉春, 任德勇 (2018). 水稻卷叶细胞学与分子机制研究进展. 植物学报 53, 848-855.
DOI |
| [14] | Akhter D (2019). 水稻叶色基因OsBML和OsPL的基因定位和功能分析. 博士论文. 杭州: 浙江大学. pp. 52-54. |
| [15] |
Chiu FY, Chen YR, Tu SL (2010). Electrostatic interaction of phytochromobilin synthase and ferredoxin for biosynthesis of phytochrome chromophore. J Biol Chem 285, 5056-5065.
DOI URL |
| [16] |
Dong H, Fei GL, Wu CY, Wu FQ, Sun YY, Chen MJ, Ren YL, Zhou KN, Cheng ZJ, Wang JL, Jiang L, Zhang X, Guo XP, Lei CL, Su N, Wang HY, Wan JM (2013). A rice virescent-yellow leaf mutant reveals new insights into the role and assembly of plastid caseinolytic protease in higher plants. Plant Physiol 162, 1867-1880.
DOI PMID |
| [17] |
Gou P, Hanke GT, Kimata-Ariga Y, Standley DM, Kubo A, Taniguchi I, Nakamura H, Hase T (2006). Higher order structure contributes to specific differences in redox potential and electron transfer efficiency of root and leaf ferredoxins. Biochemistry 45, 14389-14396.
DOI URL |
| [18] |
Jung KH, Hur J, Ryu CH, Choi Y, Chung YY, Miyao A, Hirochika H, An G (2003). Characterization of a rice chlorophyll-deficient mutant using the T-DNA gene-trap system. Plant Cell Physiol 44, 463-472.
DOI URL |
| [19] |
Li CM, Hu Y, Huang R, Ma XZ, Wang Y, Liao TT, Zhong P, Xiao FL, Sun CH, Xu ZJ, Deng XJ, Wang PR (2015). Mutation of FdC2 gene encoding a ferredoxin-like protein with C-terminal extension causes yellow-green leaf phenotype in rice. Plant Sci 238, 127-134.
DOI URL |
| [20] | Lichtenthaler HK (1987). Chlorophylls and carotenoids: pigments of photosynthetic biomembranes. Methods Enzymol 148, 350-382. |
| [21] |
Parks BM, Quail PH (1991). Phytochrome-deficient hy1 and hy2 long hypocotyl mutants of Arabidopsis are defective in phytochrome chromophore biosynthesis. Plant Cell 3, 1177-1186.
DOI URL |
| [22] |
Wang PY, Li CM, Wang Y, Huang R, Sun CH, Xu ZJ, Zhu JQ, Gao XL, Deng XJ, Wang PR (2014). Identification of a geranylgeranyl reductase gene for chlorophyll synthesis in rice. Springerplus 3, 201.
DOI URL |
| [23] |
Wu ZM, Zhang X, He B, Diao LP, Sheng SL, Wang JL, Guo XP, Su N, Wang LF, Jiang L, Wang CM, Zhai HQ, Wan JM (2007). A chlorophyll-deficient rice mutant with impaired chlorophyllide esterification in chlorophyll biosynthesis. Plant Physiol 145, 29-40.
DOI URL |
| [24] | Yoo SC, Cho SH, Sugimoto H, Li JJ, Kusumi K, Koh HJ, Iba K, Paek NC (2009). Rice virescent3 and stripe1 encoding the large and small subunits of ribonucleotide reductase are required for chloroplast biogenesis during early leaf development. Plant Physiol 150, 388-401. |
| [25] |
Zeng DC, Liu TL, Ma XL, Wang B, Zheng ZY, Zhang YL, Xie XR, Yang BW, Zhao Z, Zhu QL, Liu YG (2020a). Quantitative regulation of Waxy expression by CRISPR/ Cas9-based promoter and 5'UTR-intron editing improves grain quality in rice. Plant Biotechnol J 18, 2385-2387.
DOI URL |
| [26] |
Zeng ZQ, Lin TZ, Zhao JY, Zheng TH, Xu LF, Wang YH, Liu LL, Jiang L, Chen SH, Wan JM (2020b). OsHemA gene, encoding glutamyl-tRNA reductase (GluTR) is essential for chlorophyll biosynthesis in rice (Oryza sativa). J Integr Agricul 19, 612-623.
DOI URL |
| [27] |
Zhang HT, Li JJ, Yoo JH, Yoo SC, Cho SH, Koh HJ, Seo HS, Paek NC (2006). Rice Chlorina-1 and Chlorina-9 encode ChlD and ChlI subunits of Mg-chelatase, a key enzyme for chlorophyll synthesis and chloroplast development. Plant Mol Biol 62, 325-337.
DOI URL |
| [28] | Zhao J, Qiu ZN, Ruan BP, Kang SJ, Lei H, Zhang S, Dong GJ, Jiang H, Zeng DL, Zhang GH, Gao ZY, Ren DY, Hu XM, Chen G, Guo LB, Qian Q, Zhu L (2015). Functional inactivation of putative photosynthetic electron acceptor ferredoxin C2 (FdC2) induces delayed heading date and decreased photosynthetic rate in rice. PLoS One 10, e0143-361. |
| [29] |
Zhu XY, Guo S, Wang ZW, Du Q, Xing YD, Zhang TQ, Shen WQ, Sang XC, Ling YH, He GH (2016). Map-based cloning and functional analysis of YGL8, which controls leaf colour in rice (Oryza sativa). BMC Plant Biol 16, 134.
DOI URL |
| [1] | 叶灿, 姚林波, 金莹, 高蓉, 谭琪, 李旭映, 张艳军, 陈析丰, 马伯军, 章薇, 张可伟. 水稻水杨酸代谢突变体高通量筛选方法的建立与应用[J]. 植物学报, 2025, 60(4): 1-0. |
| [2] | 赵凌, 管菊, 梁文化, 张勇, 路凯, 赵春芳, 李余生, 张亚东. 基于高密度Bin图谱的水稻苗期耐热性QTL定位[J]. 植物学报, 2025, 60(3): 342-353. |
| [3] | 李新宇, 谷月, 徐非非, 包劲松. 水稻胚乳淀粉合成相关蛋白的翻译后修饰研究进展[J]. 植物学报, 2025, 60(2): 256-270. |
| [4] | 李建国, 张怡, 张文君. 水稻根系铁膜形成及对磷吸收的影响[J]. 植物学报, 2025, 60(1): 132-143. |
| [5] | 姚瑞枫, 谢道昕. 水稻独脚金内酯信号感知的激活和终止[J]. 植物学报, 2024, 59(6): 873-877. |
| [6] | 连锦瑾, 唐璐瑶, 张伊诺, 郑佳兴, 朱超宇, 叶语涵, 王跃星, 商文楠, 傅正浩, 徐昕璇, 吴日成, 路梅, 王长春, 饶玉春. 水稻抗氧化性状遗传位点挖掘及候选基因分析[J]. 植物学报, 2024, 59(5): 738-751. |
| [7] | 黄佳慧, 杨惠敏, 陈欣雨, 朱超宇, 江亚楠, 胡程翔, 连锦瑾, 芦涛, 路梅, 张维林, 饶玉春. 水稻突变体pe-1对弱光胁迫的响应机制[J]. 植物学报, 2024, 59(4): 574-584. |
| [8] | 周俭民. 收放自如的明星战车[J]. 植物学报, 2024, 59(3): 343-346. |
| [9] | 车佳航, 李纬楠, 秦英之, 陈金焕. 木本植物叶色变异机制研究进展[J]. 植物学报, 2024, 59(2): 319-328. |
| [10] | 朱超宇, 胡程翔, 朱哲楠, 张芷宁, 汪理海, 陈钧, 李三峰, 连锦瑾, 唐璐瑶, 钟芊芊, 殷文晶, 王跃星, 饶玉春. 水稻穗部性状QTL定位及候选基因分析[J]. 植物学报, 2024, 59(2): 217-230. |
| [11] | 夏婧, 饶玉春, 曹丹芸, 王逸, 柳林昕, 徐雅婷, 牟望舒, 薛大伟. 水稻中乙烯生物合成关键酶OsACS和OsACO调控机制研究进展[J]. 植物学报, 2024, 59(2): 291-301. |
| [12] | 王妮, 李朝娜, 郑旭理, 姜思成, 杨海芸. 花叶矢竹叶片色素合成和光合特性[J]. 植物生态学报, 2024, 48(11): 1536-1546. |
| [13] | 朱宝, 赵江哲, 张可伟, 黄鹏. 水稻细胞分裂素氧化酶9参与调控水稻叶夹角发育[J]. 植物学报, 2024, 59(1): 10-21. |
| [14] | 方妍力, 田传玉, 苏如意, 刘亚培, 王春连, 陈析丰, 郭威, 纪志远. 水稻抗细菌性条斑病基因挖掘与初定位[J]. 植物学报, 2024, 59(1): 1-9. |
| [15] | 贾绮玮, 钟芊芊, 顾育嘉, 陆天麒, 李玮, 杨帅, 朱超宇, 胡程翔, 李三峰, 王跃星, 饶玉春. 水稻茎秆细胞壁相关组分含量QTL定位及候选基因分析[J]. 植物学报, 2023, 58(6): 882-892. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||