基于CRISPR/Cas9的基因编辑技术研究进展及其在植物中的应用
何晓玲, 刘鹏程, 马伯军, 陈析丰
植物学报
2022, 57 ( 4):
508-531.
DOI: 10.11983/CBB22020
CRISPR/Cas9技术是利用RNA靶向引导Cas9核酸酶对基因组中的目标基因进行编辑的生物技术。近年来, 该技术的多种新型基因编辑器更新迅猛, 编辑效果愈加精细和高效, 在作物定向分子设计育种中展现出巨大的应用前景。该文对CRISPR/Cas9及其相关编辑器的技术原理、编辑效果和应用情况进行综述, 并探讨了该技术在应用中面临的问题、应对措施和发展前景, 旨在为相关领域的科研工作者提供参考。
View image in article
图2
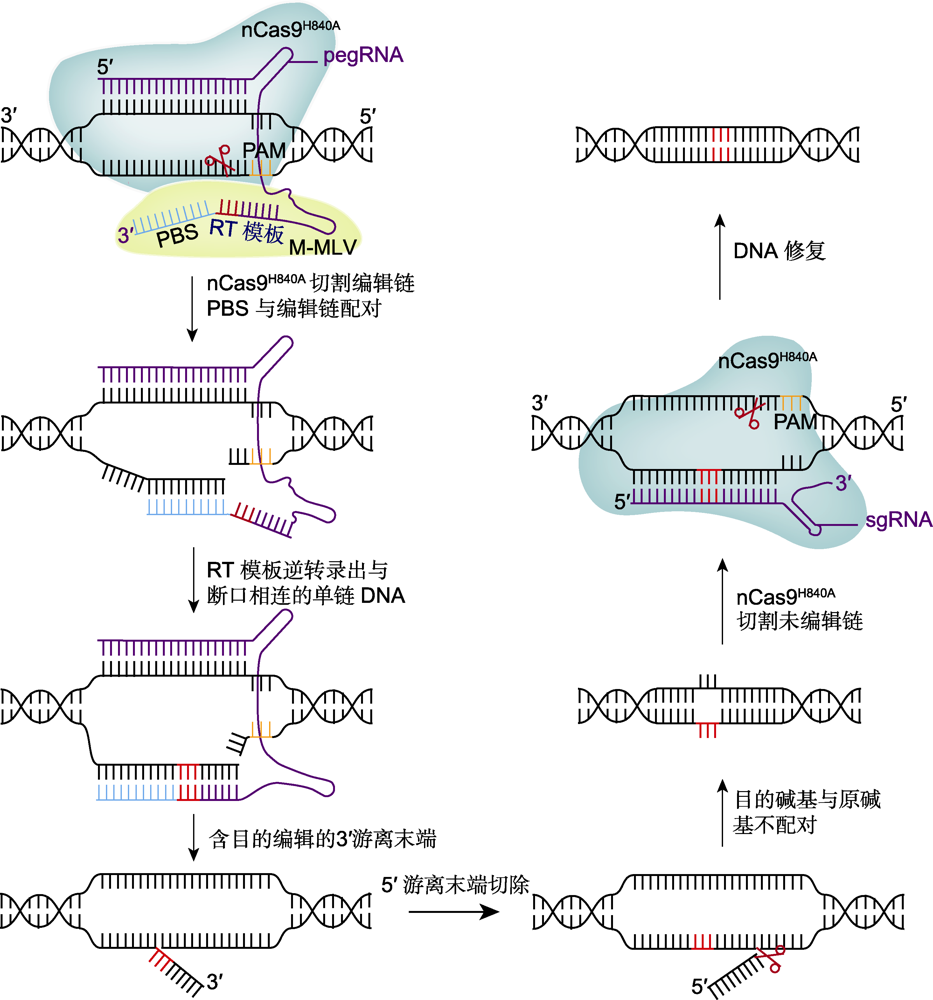
先导编辑器(PE)的工作原理
在nCas9H840A (绿)和逆转录酶M-MLV (黄)的作用下, 系统PE将碱基序列连接到靶位点, 借助DNA修复实现任意碱基的转换以及小片段的插入或删除。PAM: 原间隔序列邻近基序; PBS: 引物结合位点
正文中引用本图/表的段落
尽管增加了GBE, 与CBE和ABE一起实现了C>T、G>A、A>G、T>C、C>A、C>G、G>C和G>T 8种类型的碱基编辑, 但仍有另外4种碱基编辑类型(A>C、A>T、T>A和T>G)无法获得。为了扩展更多的编辑类型, Anzalone等(2019)研发出一种基于CRISPR/ Cas9系统的先导编辑器PE。该编辑器含有2条向导RNA: 1条特殊的pegRNA (prime editing guide RNA)以及1条简单的sgRNA。pegRNA是在sgRNA的基础上, 在3′端增加了一段具有双重功能的RNA序列, 靠近原3′末端的序列作为引物结合位点(primer binding site, PBS), 远离3′末端的序列为逆转录模板(reverse transcriptase, RT)。另一条sgRNA位于pegRNA下游约50 bp处, 且方向与其相反, 该sgRNA含有与完成编辑后的靶DNA相匹配的序列, 而与未编辑的靶序列不匹配。此外, 将Cas9蛋白HNH结构域第840位氨基酸His突变为Ala (简称H840A), 获得了仅切割sgRNA非互补链的nCas9H840A蛋白, 并在其C端融入1个M-MLV逆转录酶(含D200N/L603W/T330P/ T306K/W313F五个突变)。该编辑器工作时, nCas9H840A在pegRNA的引导下, 在靶点处切开一条ssDNA, pegRNA通过PBS序列与断开的DNA链3′末端互补配对, 并在M-MLV逆转录酶的作用下, 沿着切口3′末端, 以pegRNA的RT模板逆转录出目的ssDNA序列, 再通过DNA连接酶得到含目的序列的完整ssDNA。此时, 另一条sgRNA会靶向结合已完成编辑的目标序列, 引导nCas9H840A切割sgRNA非互补链, 再通过细胞自我修复, 以目标序列ssDNA为模板进行DNA修复, 从而获得完整的新双链DNA (图2)。PE极大地拓展了CRISPR/Cas9的基因编辑范围, 编辑位点到PAM的距离可超过30 bp, 不同于CBE和ABE受PAM的距离限制(1-20 bp)。PEs可实现12种可能的碱基替换及小片段Indel, 编辑效果也更精确(Anzalone et al., 2019; Zhu et al., 2020)。由于pegRNA的3′端增添了PBS和逆转录模板序列, 使得其3′末端过长, 容易被核酸外切酶识别并降解, 从而影响PE的编辑效率。通过在pegRNA的3′端添加特定三级结构的RNA基序(如evopreQ1或mpknot), 有助于提高pegRNA的稳定性, 抵抗其3′末端被降解, 进一步提高了PE的编辑效率(Nelson et al., 2022)。
本文的其它图/表
-
 表1
各类碱基编辑器的特点
表1
各类碱基编辑器的特点
-
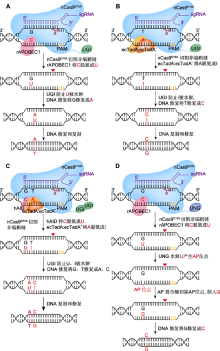 图1
单(CBE、ABE、GBE)、双(A&C-BEmax)碱基编辑器工作原理
图1
单(CBE、ABE、GBE)、双(A&C-BEmax)碱基编辑器工作原理
(A) BE3介导的C>T替换(在nCas9D10A (蓝色)、胞苷脱氨酶rAPOBEC1 (红色)和尿嘧啶糖基化酶抑制剂(UGI) (绿色)的作用下, 系统BE3将活性窗口中的C脱氨成U, 诱导细胞启动DNA修复实现C到T的替换; 红色三角形表示切口处); (B) ABE7.10介导的A>G替换(在腺苷脱氨酶ecTadA:ecTadA* (黄色和橙色)的作用下, 系统ABE7.10将活性窗口中的A脱氨成I, 并在DNA修复后实现A到G的替换); (C) A&C-BEmax介导的C>T与A>G共替换(在胞嘧啶脱氨酶hAID (红色)、ecTadA:ecTadA*和2个UGI的作用下, 系统A&C-BEmax诱导DNA修复, 实现C到T、A到G的同时替换); (D) GBE介导的C>G颠换(在尿嘧啶-N-糖基化酶(UNG) (深蓝)的作用下, UNG将C脱氨生成的U水解为AP位点, 并在DNA修复后实现C到G的颠换)。PAM: 原间隔序列邻近基序
-
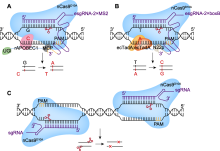 图3
多重编辑系统(SWISS)的工作原理
图3
多重编辑系统(SWISS)的工作原理
(A), (B) SWISS系统使用不同的scRNAs (MS2和boxB), 招募融合了相应蛋白(MCP和N22p)的rAPOBEC1或ecTadA:ecTadA*, 实现同时在不同位点的胞嘧啶碱基编辑(CBE)和腺嘌呤碱基编辑(ABE); (C) 使用1对sgRNA在第3个靶点产生DNA双链断裂(DSB), 诱导细胞进行同源定向修复(HDR), 产生随机突变。PAM: 原间隔序列邻近基序; UGI: 尿嘧啶糖基化酶抑制剂
-
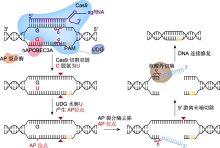 图4
多核苷酸靶向删除系统(AFID)的工作原理
图4
多核苷酸靶向删除系统(AFID)的工作原理
Cas9切割双链产生双链断裂(DSB), 尿嘧啶-DNA-糖基化酶(UDG)将C脱氨生成的U水解为AP位点, 并借助AP裂合酶(橙红色)和核酸外切酶(棕色), 实现靶点C到DSB切口之间的多核苷酸删除。PAM: 原间隔序列邻近基序
-
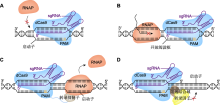 图5
CRISPR干扰系统的工作原理
图5
CRISPR干扰系统的工作原理
(A) dCas9阻止RNA聚合酶(RNAP)结合基因启动子, sgRNA介导dCas9结合目的基因启动子, 使RNAP (红色)无法与该基因启动子结合并进行转录; (B) dCas9阻断RNAP的转录延伸, sgRNA介导dCas9结合目的基因开放阅读框(ORF), 使RNAP无法继续转录延伸; (C) 转录抑制子阻止RNAP结合基因启动子, dCas9与转录抑制子(灰色)融合, 抑制子会阻止RNAP与目的基因启动子的结合; (D) 阻遏结合域(RBD)阻止目的基因的转录激活, dCas9与RBD (棕色)融合, RBD阻断转录因子(TFs)与目的基因的结合, 并与TFs结合形成强阻遏物抑制基因的转录表达。PAM: 原间隔序列邻近基序
-
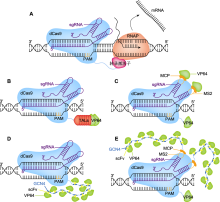 图6
CRISPR激活系统的工作原理
图6
CRISPR激活系统的工作原理
(A) 转录激活子激活目的基因转录, dCas9与转录激活子(洋红色)融合, 招募RNA聚合酶(RNAP)并激活目的基因的转录; (B)-(E) 不同CRISPRa系统示意图, 包括TALs (橙红色)、VP64 (绿色)、MS2 (浅黄色)、MCP (深黄色)、GCN4 (湛蓝色)和scFv (明黄色)。PAM: 原间隔序列邻近基序
-
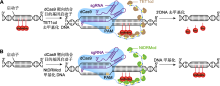 图7
基因组修饰系统去甲基化和甲基化的工作原理
图7
基因组修饰系统去甲基化和甲基化的工作原理
(A) DNA去甲基化修饰, dCas9-SunTag系统招募人脱甲基酶TET1cd (棕色), 在目的基因的启动子区使DNA去甲基化, Me表示甲基化; (B) DNA甲基化修饰, dCas9-SunTag系统招募烟草DRM甲基转移酶催化结构域NtDRMcd (绿色), 在目的基因启动子区使DNA甲基化。PAM: 原间隔序列邻近基序
-
 表2
碱基编辑器在作物中的应用
表2
碱基编辑器在作物中的应用
|
