基于CRISPR/Cas9的基因编辑技术研究进展及其在植物中的应用
何晓玲, 刘鹏程, 马伯军, 陈析丰
植物学报
2022, 57 ( 4):
508-531.
DOI: 10.11983/CBB22020
CRISPR/Cas9技术是利用RNA靶向引导Cas9核酸酶对基因组中的目标基因进行编辑的生物技术。近年来, 该技术的多种新型基因编辑器更新迅猛, 编辑效果愈加精细和高效, 在作物定向分子设计育种中展现出巨大的应用前景。该文对CRISPR/Cas9及其相关编辑器的技术原理、编辑效果和应用情况进行综述, 并探讨了该技术在应用中面临的问题、应对措施和发展前景, 旨在为相关领域的科研工作者提供参考。
编辑
技术 | 编辑器 | 脱氨酶/逆转录酶 | Cas蛋白 | PAM (5′-3′) | 活性
窗口 | 编辑类型 | 特点 | 参考文献 | 胞嘧啶碱基
编辑(CBEs) | BE3 | rAPOBEC1 | nCas9D10A | NGG | C4-C8 | C:G>T:A | 实现C>T替换, 无需DSB或提供模板 | Komor et al., 2016 | | nCas9-PBE | rAPOBEC1 | nCas9D10A | NGG | C3-C9 | 在植物中实现精准高效的C>T替换 | Zong et al., 2017 | | Target-AID | PmCDA1 | nCas9D10A | NGG | C1-C5 | 具有较好的GC碱基编辑能力 | Nishida et al., 2016 | | dCas9-AIDx | hAID | dCas9 | NCN | C4-C9 | Ma et al., 2016 | A3AY130F-
CBE_V01
A3AY130F-
CBE_V04 | hAPOBEC3AY130F | nCas9D10A | NGG | C4-C15 | 活性窗口拓宽至12 nt | Ren et al., 2021a | | eCDAL | LjCDA1L-4 | nCas9D10A | NGG | C1-C12 | Xu et al., 2021 | | BE3-PAPAPAP | rAPOBEC1 | nCas9D10A | NGG | C5-C6 | 活性窗口缩小至1-2 nt | Tan et al., 2019 | | PhieCBEs | evorAPOCBEC1
evoFERNY
evoCDA1
hA3A | nCas9-NG
eCas9n-
NG | NG
NGG | C-1-C15 | 编辑窗口广 | Zeng et al., 2020 | BE3R126E
BE3R132E
YE1-BE3 | rAPOBEC1R126E
rAPOBEC1R132E
rAPOBEC1W90Y+R126E | nCas9D10A | NGG | C5-C7 | 脱靶率显著降低 | Zuo et al., 2020 | 腺嘌呤碱基
编辑(ABEs) | ABE7.10 | ecTadA:ecTadA* | nCas9D10A | NGG | A4-A7 | A:T>G:C | 实现A>G替换, 无需DSB或提供模板 | Gaudelli et al., 2017 | | PABE | ecTadA:ecTadA* | nCas9D10A | NGG | A4-A8 | 在植物中实现精准高效的A>G替换 | Li et al., 2018b | | ABE-Ps | ecTadA:ecTadA* | nCas9D10A
SaCas9 | NGG
NNNRRT | A1-A15 | Hua et al., 2018 | | rBE14 | ecTadA:ecTadA* | nCas9D10A | NGG | A5-A7 | 荧光检测编辑植株 | Yan et al., 2018 | | rABE8e | TadA8eV106W | nCas9D10A
nCas9-NG | NG
NGG | A5-A6 | 极高的靶点编辑效率和碱基纯合替换效率 | Wei et al., 2021 | | PhieABEs | TadA8e | nCas9-NG
SpGn
SpRYn | NG
NGN
NNN | A1-A14 | 编辑效率高, 几近无PAM靶向, 窗口广 | Tan et al., 2022 | | SpRY-ABE8e | ecTadA:ecTadA* | nCas9D10A | NNN | A3-A10 | 几近无PAM靶向 | Ren et al., 2021b | | ABE7.10F148A | ecTadAF148A: ecTadA*F148A | nCas9D10A | NGG | A5-A6 | 活性窗口缩小至1-2 nt | Zhou et al., 2019 | | CG碱基编辑(GBEs) | GBE | AID
rAPOBEC1 | nCas9D10A | NGG | C3-C7 | C:G>A:T/G:C | 实现嘧啶与嘌呤间的颠换 | Zhao et al., 2021 | | CGBE | rAPOBEC1 | C5-C6 | Chen et al., 2021 | | 双碱基编辑(A&CBE) | A&C-BEmax | hAID
ecTadA:ecTadA* | nCas9D10A | NGG | C2-C17
A4-A7 | C:G>T:A
A:T>G:C | 实现A、C共编辑 | Zhang et al., 2020 | | Target-ACE | PmCDA1
ecTadA:ecTadA* | NGG | C1-C10
A4-A8 | Sakata et al., 2019 | | SPACE | PmCDA1
ecTadA* | NGG | C2-C7
A4-A7 | Grünewald et al., 2020 | | ACBE | PmCDA1
ecTadA:ecTadA* | NGG | C1-C7
A4-A6 | Xie et al., 2020 | | STEME | hAPOBEC3A
ecTadA:ecTadA* | NG
NGD | C1-C17
A4-A8 | 在植物中实现A、
C共编辑 | Li et al., 2020a | | pDuBE1 | TadA8e | NGG | C1-C10
A2-A9 | Xu et al., 2021 | 编辑
技术 | 编辑器 | 脱氨酶/逆转录酶 | Cas蛋白 | PAM (5′-3′) | 活性
窗口 | 编辑类型 | 特点 | 参考文献 | | 先导编辑(PEs) | PE | M-MLV RT | nCas9H840A | NGG | 1-50 | 12种碱基替换
插入(<15 bp)
缺失(<40 bp) | 不受PAM的距离
限制 | Anzalone et al., 2019 | | PPE | CaMV RT
Retron RT | 在植物中实现PE
应用 | Lin et al., 2020 | | pPE2 | M-MLV RT | Xu et al., 2020a | | ePPE | M-MLV RT∆RNase H
M-MLV RT:NC | 1-92 | 12种碱基替换
插入(<40 bp)
缺失(<100 bp) | 提高了在植物中的
编辑效率 | Zong et al., 2022 | 多重
编辑 | SWISSs | rAPOBEC1
ecTadA:ecTadA* | nCas9D10A | NG | C3-C16
A4-A7 | C:G>T:A
A:T>G:C
Indels | 具有A>G、C>T和Indels三重编辑功能 | Li et al., 2020b | 片段删
除编辑 | AFIDs | hAPOBEC3A
hAPOBEC3Bctd | Cas9 | NGG | C1-C14 | 多核苷酸删除 | 精准高效、可预测的多核苷酸缺失 | Wang et al., 2020 |
View table in article
表1
各类碱基编辑器的特点
正文中引用本图/表的段落
由于CRISPR/Cas9需要产生DSB进行基因编辑, 因此具有一定的局限性, 因为NHEJ产生的突变是不定向的, 无法高效地获得预期的突变, 而HDR虽然可借助“人工模板”进行定向突变, 但其编辑效率非常低(Symington and Gautier, 2011)。因此, CRISPR/ Cas9技术在基因敲除中能高效应用, 只需造成基因移码突变就能破坏其编码蛋白的功能, 但对基因序列的定向改造或插入预设序列的效果并不理想, 大大限制了其应用范围。于是, 研究人员不断地对CRISPR/ Cas9系统进行改造, 如对Cas9蛋白的功能结构域进行突变, 引入不同类型的碱基脱氨酶, 开发出各类碱基编辑器(base editor, BE), 如胞嘧啶碱基编辑(cytidine BE, CBE)、腺嘌呤碱基编辑(adenine BE, ABE)和CG碱基编辑器(C-to-G BE, GBE) (表1), 可以对PAM 5'端的原间隔序列范围内的4- -10 nt (也称编辑活性窗口(activity windows))进行有效的碱基替换(substitution)或颠换(transversion), BE的定向突变率是HDR的10-100倍(Yeh et al., 2018)。同时, 为了弥补BE的碱基编辑类型有限以及不能引入预期的小片段插入或缺失(insertion or deletion, Indel), 还开发出先导编辑(prime editor, PE)、多重编辑(simultaneous and wide-editing induced by a single system, SWISS)和片段删除编辑(APOBEC-Cas9 fusion-induced deletion system, AFID)等新技术(表1), 从而实现对靶基因高效率、高精度和高需求的编辑。
N表示C、A、G、T四种碱基. ... In vivo base editing of post-mitotic sensory cells 1 2018 ... 由于CRISPR/Cas9需要产生DSB进行基因编辑, 因此具有一定的局限性, 因为NHEJ产生的突变是不定向的, 无法高效地获得预期的突变, 而HDR虽然可借助“人工模板”进行定向突变, 但其编辑效率非常低(Symington and Gautier, 2011).因此, CRISPR/ Cas9技术在基因敲除中能高效应用, 只需造成基因移码突变就能破坏其编码蛋白的功能, 但对基因序列的定向改造或插入预设序列的效果并不理想, 大大限制了其应用范围.于是, 研究人员不断地对CRISPR/ Cas9系统进行改造, 如对Cas9蛋白的功能结构域进行突变, 引入不同类型的碱基脱氨酶, 开发出各类碱基编辑器(base editor, BE), 如胞嘧啶碱基编辑(cytidine BE, CBE)、腺嘌呤碱基编辑(adenine BE, ABE)和CG碱基编辑器(C-to-G BE, GBE) (表1), 可以对PAM 5'端的原间隔序列范围内的4- -10 nt (也称编辑活性窗口(activity windows))进行有效的碱基替换(substitution)或颠换(transversion), BE的定向突变率是HDR的10-100倍(Yeh et al., 2018).同时, 为了弥补BE的碱基编辑类型有限以及不能引入预期的小片段插入或缺失(insertion or deletion, Indel), 还开发出先导编辑(prime editor, PE)、多重编辑(simultaneous and wide-editing induced by a single system, SWISS)和片段删除编辑(APOBEC-Cas9 fusion-induced deletion system, AFID)等新技术(表1), 从而实现对靶基因高效率、高精度和高需求的编辑. ... PhieCBEs: plant high-efficiency cytidine base editors with expanded target range 2 2020 ... Characteristics of all kinds of base editors
本文的其它图/表
-
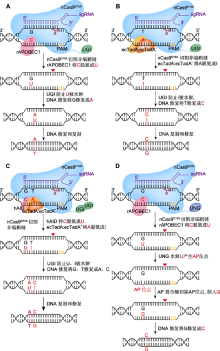 图1
单(CBE、ABE、GBE)、双(A&C-BEmax)碱基编辑器工作原理
图1
单(CBE、ABE、GBE)、双(A&C-BEmax)碱基编辑器工作原理
(A) BE3介导的C>T替换(在nCas9D10A (蓝色)、胞苷脱氨酶rAPOBEC1 (红色)和尿嘧啶糖基化酶抑制剂(UGI) (绿色)的作用下, 系统BE3将活性窗口中的C脱氨成U, 诱导细胞启动DNA修复实现C到T的替换; 红色三角形表示切口处); (B) ABE7.10介导的A>G替换(在腺苷脱氨酶ecTadA:ecTadA* (黄色和橙色)的作用下, 系统ABE7.10将活性窗口中的A脱氨成I, 并在DNA修复后实现A到G的替换); (C) A&C-BEmax介导的C>T与A>G共替换(在胞嘧啶脱氨酶hAID (红色)、ecTadA:ecTadA*和2个UGI的作用下, 系统A&C-BEmax诱导DNA修复, 实现C到T、A到G的同时替换); (D) GBE介导的C>G颠换(在尿嘧啶-N-糖基化酶(UNG) (深蓝)的作用下, UNG将C脱氨生成的U水解为AP位点, 并在DNA修复后实现C到G的颠换)。PAM: 原间隔序列邻近基序
-
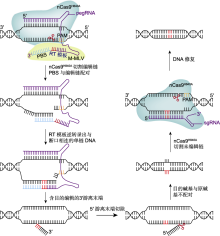 图2
先导编辑器(PE)的工作原理
图2
先导编辑器(PE)的工作原理
在nCas9H840A (绿)和逆转录酶M-MLV (黄)的作用下, 系统PE将碱基序列连接到靶位点, 借助DNA修复实现任意碱基的转换以及小片段的插入或删除。PAM: 原间隔序列邻近基序; PBS: 引物结合位点
-
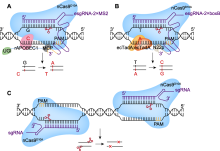 图3
多重编辑系统(SWISS)的工作原理
图3
多重编辑系统(SWISS)的工作原理
(A), (B) SWISS系统使用不同的scRNAs (MS2和boxB), 招募融合了相应蛋白(MCP和N22p)的rAPOBEC1或ecTadA:ecTadA*, 实现同时在不同位点的胞嘧啶碱基编辑(CBE)和腺嘌呤碱基编辑(ABE); (C) 使用1对sgRNA在第3个靶点产生DNA双链断裂(DSB), 诱导细胞进行同源定向修复(HDR), 产生随机突变。PAM: 原间隔序列邻近基序; UGI: 尿嘧啶糖基化酶抑制剂
-
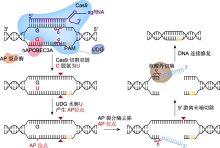 图4
多核苷酸靶向删除系统(AFID)的工作原理
图4
多核苷酸靶向删除系统(AFID)的工作原理
Cas9切割双链产生双链断裂(DSB), 尿嘧啶-DNA-糖基化酶(UDG)将C脱氨生成的U水解为AP位点, 并借助AP裂合酶(橙红色)和核酸外切酶(棕色), 实现靶点C到DSB切口之间的多核苷酸删除。PAM: 原间隔序列邻近基序
-
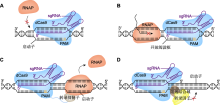 图5
CRISPR干扰系统的工作原理
图5
CRISPR干扰系统的工作原理
(A) dCas9阻止RNA聚合酶(RNAP)结合基因启动子, sgRNA介导dCas9结合目的基因启动子, 使RNAP (红色)无法与该基因启动子结合并进行转录; (B) dCas9阻断RNAP的转录延伸, sgRNA介导dCas9结合目的基因开放阅读框(ORF), 使RNAP无法继续转录延伸; (C) 转录抑制子阻止RNAP结合基因启动子, dCas9与转录抑制子(灰色)融合, 抑制子会阻止RNAP与目的基因启动子的结合; (D) 阻遏结合域(RBD)阻止目的基因的转录激活, dCas9与RBD (棕色)融合, RBD阻断转录因子(TFs)与目的基因的结合, 并与TFs结合形成强阻遏物抑制基因的转录表达。PAM: 原间隔序列邻近基序
-
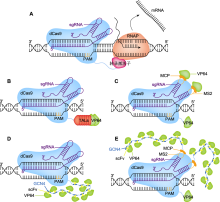 图6
CRISPR激活系统的工作原理
图6
CRISPR激活系统的工作原理
(A) 转录激活子激活目的基因转录, dCas9与转录激活子(洋红色)融合, 招募RNA聚合酶(RNAP)并激活目的基因的转录; (B)-(E) 不同CRISPRa系统示意图, 包括TALs (橙红色)、VP64 (绿色)、MS2 (浅黄色)、MCP (深黄色)、GCN4 (湛蓝色)和scFv (明黄色)。PAM: 原间隔序列邻近基序
-
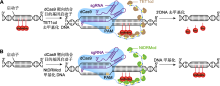 图7
基因组修饰系统去甲基化和甲基化的工作原理
图7
基因组修饰系统去甲基化和甲基化的工作原理
(A) DNA去甲基化修饰, dCas9-SunTag系统招募人脱甲基酶TET1cd (棕色), 在目的基因的启动子区使DNA去甲基化, Me表示甲基化; (B) DNA甲基化修饰, dCas9-SunTag系统招募烟草DRM甲基转移酶催化结构域NtDRMcd (绿色), 在目的基因启动子区使DNA甲基化。PAM: 原间隔序列邻近基序
-
 表2
碱基编辑器在作物中的应用
表2
碱基编辑器在作物中的应用
|
