基于CRISPR/Cas9的基因编辑技术研究进展及其在植物中的应用
何晓玲, 刘鹏程, 马伯军, 陈析丰
植物学报
2022, 57 ( 4):
508-531.
DOI: 10.11983/CBB22020
CRISPR/Cas9技术是利用RNA靶向引导Cas9核酸酶对基因组中的目标基因进行编辑的生物技术。近年来, 该技术的多种新型基因编辑器更新迅猛, 编辑效果愈加精细和高效, 在作物定向分子设计育种中展现出巨大的应用前景。该文对CRISPR/Cas9及其相关编辑器的技术原理、编辑效果和应用情况进行综述, 并探讨了该技术在应用中面临的问题、应对措施和发展前景, 旨在为相关领域的科研工作者提供参考。
| 物种 | 品种 | 靶基因 | 转化方法 | 编辑
技术 | 脱氨酶/逆转录酶 | 编辑类型 | 编辑
窗口 | 编辑效率(%) | 参考文献 | | 水稻(Oryza sativa) | 中花11 | CDC48
NRT1.1B-T1 | 农杆菌AGL1 | CBEs | hAPOBEC3A | C:G>T:A | C2-C16 | 44.1-
82.9 | Zong et al., 2018 | | 日本晴 | CDC48 | 农杆菌AGL1 | rAPOBEC1 | C:G>T:A | C3-C8 | 43.48 | Zong et al., 2017 | | Kitaake | Pi-d2
FLS2 | 农杆菌EHA105 | hAID*Δ | C:G>T:A | C3-C7 | 30.8-57 | Ren et al., 2018 | | 中花11 | ACC-T1
ALS-T1
CDC48-T3
DEP1-T1/2
NRT1.1B-T1 | 农杆菌AGL1 | ABEs | ecTadA:
ecTadA* | A:T>G:C | A4-A8 | 15.8-
59.1 | Li et al., 2018b | | 日本晴 | SPL14/16/17/18
SLR1 | 农杆菌EHA105 | ecTadA:
ecTadA* | A:T>G:C | A5-A14 | 12.5-
61.3 | Hua et al., 2018 | | Kitaake | SERK2
WRKY45 | 农杆菌EHA105 | ecTadA:
ecTadA* | A:T>G:C | A4-A7 | 32.05-
62.26 | Yan et al., 2018 | | 中花11 | ACC | 农杆菌AGL1 | STEMEs | hAPOBEC3A
ecTadA:
ecTadA* | C:G>T:A; A:T>G:C | C1-C17
A4-A8 | 3.84 | Li et al., 2020a | | 中花11 | CDC48-T1
ALS-T2 | 农杆菌EHA105 | PEs | M-MLV | G:C>T:A
插入(≤3 bp)
删除(≤6 bp) | N1-N6 | 2.6-21.8 | Lin et al., 2020 | | 日本晴 | ALS-1/2
ACC-1
DEP1 | 农杆菌EHA105 | PEs | M-MLV | C:G>T:A
A:T>G:C
G:C>T:A
G:C>C:G
G:C>A:T
T:A>A:T
A:T>C:G | N18-N33 | 1.7-26 | Xu et al., 2020b | | 物种 | 品种 | 靶基因 | 转化方法 | 编辑
技术 | 脱氨酶/逆转录酶 | 编辑类型 | 编辑
窗口 | 编辑效率(%) | 参考文献 | | 中花11 | ALS-T2
ACC-T2
BADH-indels | 农杆菌AGL1 | SWISS | hAPOBEC3A
ecTadA:
ecTadA* | C:G>T:A
A:T>G:C
C:G>G:C
删除(≤45 bp) | C6-C7
A4-A7 | 7.3 | Li et al., 2020b | | 中花11 | CDC48-T2
SPL14
SWEET14 | 农杆菌AGL1 | AFID | hAPOBEC3A | 删除(≤16 bp) | C2-C17 | 22.2-
55.8 | Wang et al., 2020 | | 小麦(Triticum aestivum) | Kenong 199 | ALS
MTL | 基因枪 | CBEs | hAPOBEC3A | C:G>T:A | C-9-C13 | 16.7-
22.5 | Zong et al., 2018 | | Bobwhite | LOX2-S1 | 基因枪 | CBEs | rAPOBEC1 | C:G>T:A | C3-C9 | 1.25 | Zong et al., 2017 | | Kenong 199 | ALS | 基因枪 | CBEs | rAPOBEC1 | C:G>T:A | C-1-C7 | 22-78 | Zhang et al., 2019 | | Kenong 199 | DEP1
GW2 | 基因枪 | ABEs | ecTadA:
ecTadA* | A:T>G:C | A5-A8 | 0.4-1.1 | Li et al., 2018a | | Kenong 199 | miR396
GASR6 | 基因枪 | AFID | hAPOBEC3A | 删除(≤35 bp) | C-12-C23 | 25-37.5 | Wang et al., 2020 | | 番茄(Solanum lycopersicum) | WVA106 | ALS1 | 农杆菌C58 pGV2260 | CBEs | PmCDA1 | C:G>T:A | C7 | 34.7 | Veillet et al., 2019 | | WVA106 | ALS1 | 农杆菌C58 pGV2260 | CBEs | PmCDA1 | C:G>T:A | C1-C8 | 20.59 | Veillet et al., 2020 | | Micro- Tom | DELLA | 农杆菌 | CBEs | PmCDA1 | C:G>T:A | C1-C3 | 50.5 | Shimatani et al., 2017 | 马铃薯
(S. tuberosum) | Désirée | GBSS1
DMR6-1 | PEG | CBEs | hAPOBEC3A | C:G>T:A | C3-C10 | 8-16.67 | Veillet et al., 2020 | | Désirée | GBSS-T6 | PEG | CBEs | hAPOBEC3A | C:G>T:A | C1-C13 | 6.5 | Jiang et al., 2022 | | Désirée | ALS1 | 农杆菌C58 pGV2260 | CBEs | PmCDA1 | C:G>T:A | C1-C8 | 25 | Veillet et al., 2019 | 大豆
(Glycine max) | Jack | FT2a
FT4 | 农杆菌 | CBEs | rAPOBEC1 | C:G>T:A | C6-C7 | 5.88-
18.2 | Cai et al., 2020 | | 棉花(Gossypium hirsutum) | Jin668 | CLA
PEBP | 农杆菌GV3101 | CBEs | rAPOBEC1 | C:G>T:A | C4-C8 | 26.67-
57.78 | Qin et al., 2020 | 玉米
(Zea mays) | Zong31 | CENH3 | 农杆菌AGL1 | CBEs | rAPOBEC1 | C:G>T:A | C3-C8 | 10.1 | Zong et al., 2017 | | ND73 | ALS1/2 | 农杆菌LBA4404/ pVS1-VIR2 | PEs | CmYLCV | C:G>T:A
A:T>C:G
T:A>G:C
T:A>C:G
C:G>A:T
G:C>T:A
G:C>A:T
G:C>C:G | N3-N46 | 6.5-53.2 | Jiang et al., 2020 | | 油菜(Brassica napus) | J9712 | ALS1 | 农杆菌 | CBEs | rAPOBEC1 | C:G>T:A | C5-C7 | 1.8 | Wu et al., 2020 | | 西瓜(Citrullus lanatus) | ZG94 | ALS | 农杆菌EHA105 | CBEs | rAPOBEC1 | C:G>T:A | C7-C8 | 23 | Tian et al., 2018 |
View table in article
表2
碱基编辑器在作物中的应用
正文中引用本图/表的段落
CRISPR/Cas9基因编辑技术自诞生以来, 凭借其无与伦比的编辑优势, 迅速成为生物学领域中的强大工具。相较于作物的传统育种方式, 基因编辑育种具有精准、高效、成本低和周期短等多方面优点, 且其本质上类似于辐射或化学诱变育种, 但又能实现定向设计的分子育种。目前已成功在水稻、小麦、番茄(Solanum lycopersicum)、玉米、大豆(Glycine max)和甘蓝型油菜(Brassica napus)等多种作物中取得了较好的应用(表2)。CRISPR/Cas9基因编辑技术以前所未有的速度和效率打破了传统育种的瓶颈, 它凭借精准的编辑引入功能性突变, 删除产生不良性状的基因序列, 从而改良作物品质, 增强作物抗病能力, 提高其抗逆性与产量, 甚至可对野生种质资源进行从头驯化(de novo domestication) (谭禄宾和孙传清, 2021)。
然而, 直接敲除易感基因会导致植物损失部分内源功能基因, 影响植物的正常生长, 因此通过敲除易感基因使植株获得抗病能力并不是最理想的方式。而利用BEs对基因进行单碱基编辑, 可以在避免敲除基因的同时, 介导功能基因的表达变化, 例如, 通过BEs介导水稻FLS2基因的单碱基替换, 对其Ala948位点进行Asp或Glu替换, 使FLS2激酶结构域发生改变, 能促使水稻对病原性细菌产生更强的免疫反应; 同样通过单碱基突变, 还可以介导显隐性抗病基因之间的转化, 例如, 在水稻隐性基因pi-d2第441位氨基酸上引入G>A突变(M441I), 使其转化为显性抗性等位基因Pi-d2, 从而恢复其稻瘟病R基因的生物学功能(Ren et al., 2018)。
本文的其它图/表
-
 表1
各类碱基编辑器的特点
表1
各类碱基编辑器的特点
-
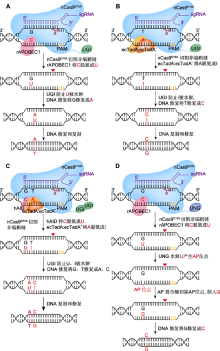 图1
单(CBE、ABE、GBE)、双(A&C-BEmax)碱基编辑器工作原理
图1
单(CBE、ABE、GBE)、双(A&C-BEmax)碱基编辑器工作原理
(A) BE3介导的C>T替换(在nCas9D10A (蓝色)、胞苷脱氨酶rAPOBEC1 (红色)和尿嘧啶糖基化酶抑制剂(UGI) (绿色)的作用下, 系统BE3将活性窗口中的C脱氨成U, 诱导细胞启动DNA修复实现C到T的替换; 红色三角形表示切口处); (B) ABE7.10介导的A>G替换(在腺苷脱氨酶ecTadA:ecTadA* (黄色和橙色)的作用下, 系统ABE7.10将活性窗口中的A脱氨成I, 并在DNA修复后实现A到G的替换); (C) A&C-BEmax介导的C>T与A>G共替换(在胞嘧啶脱氨酶hAID (红色)、ecTadA:ecTadA*和2个UGI的作用下, 系统A&C-BEmax诱导DNA修复, 实现C到T、A到G的同时替换); (D) GBE介导的C>G颠换(在尿嘧啶-N-糖基化酶(UNG) (深蓝)的作用下, UNG将C脱氨生成的U水解为AP位点, 并在DNA修复后实现C到G的颠换)。PAM: 原间隔序列邻近基序
-
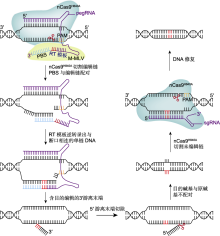 图2
先导编辑器(PE)的工作原理
图2
先导编辑器(PE)的工作原理
在nCas9H840A (绿)和逆转录酶M-MLV (黄)的作用下, 系统PE将碱基序列连接到靶位点, 借助DNA修复实现任意碱基的转换以及小片段的插入或删除。PAM: 原间隔序列邻近基序; PBS: 引物结合位点
-
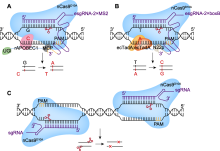 图3
多重编辑系统(SWISS)的工作原理
图3
多重编辑系统(SWISS)的工作原理
(A), (B) SWISS系统使用不同的scRNAs (MS2和boxB), 招募融合了相应蛋白(MCP和N22p)的rAPOBEC1或ecTadA:ecTadA*, 实现同时在不同位点的胞嘧啶碱基编辑(CBE)和腺嘌呤碱基编辑(ABE); (C) 使用1对sgRNA在第3个靶点产生DNA双链断裂(DSB), 诱导细胞进行同源定向修复(HDR), 产生随机突变。PAM: 原间隔序列邻近基序; UGI: 尿嘧啶糖基化酶抑制剂
-
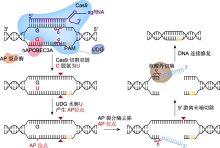 图4
多核苷酸靶向删除系统(AFID)的工作原理
图4
多核苷酸靶向删除系统(AFID)的工作原理
Cas9切割双链产生双链断裂(DSB), 尿嘧啶-DNA-糖基化酶(UDG)将C脱氨生成的U水解为AP位点, 并借助AP裂合酶(橙红色)和核酸外切酶(棕色), 实现靶点C到DSB切口之间的多核苷酸删除。PAM: 原间隔序列邻近基序
-
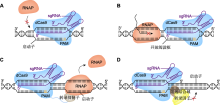 图5
CRISPR干扰系统的工作原理
图5
CRISPR干扰系统的工作原理
(A) dCas9阻止RNA聚合酶(RNAP)结合基因启动子, sgRNA介导dCas9结合目的基因启动子, 使RNAP (红色)无法与该基因启动子结合并进行转录; (B) dCas9阻断RNAP的转录延伸, sgRNA介导dCas9结合目的基因开放阅读框(ORF), 使RNAP无法继续转录延伸; (C) 转录抑制子阻止RNAP结合基因启动子, dCas9与转录抑制子(灰色)融合, 抑制子会阻止RNAP与目的基因启动子的结合; (D) 阻遏结合域(RBD)阻止目的基因的转录激活, dCas9与RBD (棕色)融合, RBD阻断转录因子(TFs)与目的基因的结合, 并与TFs结合形成强阻遏物抑制基因的转录表达。PAM: 原间隔序列邻近基序
-
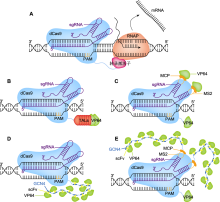 图6
CRISPR激活系统的工作原理
图6
CRISPR激活系统的工作原理
(A) 转录激活子激活目的基因转录, dCas9与转录激活子(洋红色)融合, 招募RNA聚合酶(RNAP)并激活目的基因的转录; (B)-(E) 不同CRISPRa系统示意图, 包括TALs (橙红色)、VP64 (绿色)、MS2 (浅黄色)、MCP (深黄色)、GCN4 (湛蓝色)和scFv (明黄色)。PAM: 原间隔序列邻近基序
-
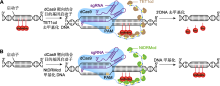 图7
基因组修饰系统去甲基化和甲基化的工作原理
图7
基因组修饰系统去甲基化和甲基化的工作原理
(A) DNA去甲基化修饰, dCas9-SunTag系统招募人脱甲基酶TET1cd (棕色), 在目的基因的启动子区使DNA去甲基化, Me表示甲基化; (B) DNA甲基化修饰, dCas9-SunTag系统招募烟草DRM甲基转移酶催化结构域NtDRMcd (绿色), 在目的基因启动子区使DNA甲基化。PAM: 原间隔序列邻近基序
|
