

植物学报 ›› 2023, Vol. 58 ›› Issue (1): 159-168.DOI: 10.11983/CBB22188 cstr: 32102.14.CBB22188
所属专题: 杂粮生物学专辑 (2023年58卷1期)
• 研究报告 • 上一篇
王宇卓1, 林元香1, 薛亚鹏1, 段政勇1, 王晓丹1, 陈凌2, 曹晓宁2, 王瑞云1,2,*( ), 乔治军2,*(
), 乔治军2,*( )
)
收稿日期:2022-08-06
接受日期:2023-01-10
出版日期:2023-01-01
发布日期:2023-01-10
通讯作者:
*E-mail: wry925@126.com;nkypzs@126.com
基金资助:
Yuzhuo Wang1, Yuanxiang Lin1, Yapeng Xue1, Zhengyong Duan1, Xiaodan Wang1, Ling Chen2, Xiaoning Cao2, Ruiyun Wang1,2,*( ), Zhijun Qiao2,*(
), Zhijun Qiao2,*( )
)
Received:2022-08-06
Accepted:2023-01-10
Online:2023-01-01
Published:2023-01-10
Contact:
*E-mail: wry925@126.com;nkypzs@126.com
摘要: 为快速鉴定糜子(Panicum miliaceum)资源, 建立分子标记检测平台, 以272份山西糜子核心种质为研究材料, 利用85对简单重复序列(SSR)引物, 应用ID Analysis 4.0软件, 构建DNA分子身份证。结果表明, 对85对SSR引物进行筛选, 发现20对引物组合(RYW67、RYW53、RYW37、RYW65、RYW62、RYW77、RYW5、RYW49、RYW84、RYW19、RYW11、RYW40、RYW54、RYW28、RYW31、RYW7、RYW16、RYW8、RYW9和RYW18)可区分272份材料。共检测到等位变异(Na) 60个, 平均每个位点检出3个; Shannon多样性指数(I)为0.957 8 (RYW16)-1.096 7 (RYW5), 平均值为1.055 2; 多态性信息含量(PIC)为0.604 4 (RYW77)-0.753 0 (RYW37), 平均值为0.692 1。利用20对引物构建山西糜子核心种质的字符串、条形码和二维码DNA分子身份证, 可为种质身份标识和溯源提供实践路径。
王宇卓, 林元香, 薛亚鹏, 段政勇, 王晓丹, 陈凌, 曹晓宁, 王瑞云, 乔治军. 山西糜子核心种质分子身份证构建. 植物学报, 2023, 58(1): 159-168.
Yuzhuo Wang, Yuanxiang Lin, Yapeng Xue, Zhengyong Duan, Xiaodan Wang, Ling Chen, Xiaoning Cao, Ruiyun Wang, Zhijun Qiao. Construction of Molecular ID Card of Core Germplasm of Hog Millet (Panicum miliaceum) in Shanxi. Chinese Bulletin of Botany, 2023, 58(1): 159-168.
| Number | Unicode | Name | Origin | Remark |
|---|---|---|---|---|
| 1 | Jinshu 5 | Datong | Bred variety | |
| 2 | Baiyingmi | Dingxiang, Xinzhou | Farmer | |
| 3 | Yingshu | Gaoping, Jincheng | Farmer | |
| 4 | 00001271 | Heishu | Pingding, Yangquan | Landrace |
| 5 | 00001352 | Chengshuhong | Fenyang, Lüliang | Landrace |
| 6 | 00003199 | Huangmizi | Huairen, Datong | Landrace |
表1 用于SSR引物初步筛选的糜子资源
Table 1 Hog millet accessions for preliminary screening of SSR primers
| Number | Unicode | Name | Origin | Remark |
|---|---|---|---|---|
| 1 | Jinshu 5 | Datong | Bred variety | |
| 2 | Baiyingmi | Dingxiang, Xinzhou | Farmer | |
| 3 | Yingshu | Gaoping, Jincheng | Farmer | |
| 4 | 00001271 | Heishu | Pingding, Yangquan | Landrace |
| 5 | 00001352 | Chengshuhong | Fenyang, Lüliang | Landrace |
| 6 | 00003199 | Huangmizi | Huairen, Datong | Landrace |
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) | Tm (°C) | Repeat motif |
|---|---|---|---|---|
| RYW5 | GACGATGCTCTTGACCTTGT | CACCGTGAAATGTCTCTGCT | 55.00 | (CCTTT)5 |
| RYW7 | TCCACTCATCCATTGCTCGT | GATGGATTCAAAGGGACGCT | 55.65 | (CGCGC)5 |
| RYW8 | GGGTCAGAGAATACACAGCG | GTAGGGAAGGAGAAGTGGGT | 55.90 | (AATAG)5 |
| RYW9 | GGACCCTTCCCTCACAGATT | TCCAGTTGCTCTTGCCGTT | 57.10 | (CTAG)6 |
| RYW11 | TGCTCGTCTTCTCGCTTCG | AGTAGTCCTCCACCGCCATCT | 59.20 | (GGTA)5 |
| RYW16 | ATCTCCTCCGCCTTCTAACCC | TGGCAATGGTCGTACAAACT | 56.65 | (GAGC)5 |
| RYW18 | CTCCCTCTTTGTCCTCGTT | GCTGCCTCTTCGCTATCTT | 54.45 | (AGTT)6 |
| RYW19 | GAATGATAGGTCCGCAAGG | CAGCCTTTGTTCAGTTGTCTC | 53.45 | (TTAT)5 |
| RYW28 | CCAAGGCTGAGCAGAAAGAT | ACAAGGTGAAACCCGAAGC | 55.40 | (AGGC)5 |
| RYW31 | ACCCAGAGTCCAGAGAAGC | GATGTCCTCCTCCTTCTCC | 55.60 | (AGCG)5 |
| RYW37 | CATTCCGTTCCTTGTCTTCC | CAGTCTCACTCCTGCGATGT | 55.30 | (GCGAT)5 |
| RYW40 | TGCTCTTCGGCTCTTCTCC | ATCAGCTCATCGTGACCCC | 57.35 | (CAGC)6 |
| RYW49 | GCTAAATCCGCTGATGAGGT | TGTATGTTGCTCCAGCCTTG | 55.05 | (TATC)6 |
| RYW53 | ATGCCTCCGATGTAGATGC | GCCGCCTTCTCTTCATTCT | 54.90 | (GAGG)5 |
| RYW54 | GCACTTGCTCCTGCTTCTC | GACCTTGCCGATGTTGTTG | 55.65 | (CCTC)5 |
| RYW62 | GTTTAGAGAGCAGGAGGCG | AGCCCTGTCCACCCTAATC | 51.40 | (GCTC)5 |
| RYW65 | TAGCGTCGTCAAGAAGCACT | ACAGAGGTGACGAAACGATG | 55.40 | (GCCG)5 |
| RYW67 | GAAGGAAACGCACCAGAGT | TTGGGTTTGTGCTTGGAGT | 55.25 | (TGCG)5 |
| RYW77 | CAGCAAGCAGACAGAGCAG | ACAAAGCAGAGAGGGGAAG | 55.65 | (ATAC)5 |
| RYW84 | AACCCACCCATCCATTCCT | TAAGTCGTCGTCGGCAGAGT | 57.35 | (GAGC)5 |
表2 用于构建分子身份证的20对SSR引物
Table 2 Twenty pairs of SSR primers used to construct molecular ID cards
| Primer name | Forward primer (5′-3′) | Reverse primer (5′-3′) | Tm (°C) | Repeat motif |
|---|---|---|---|---|
| RYW5 | GACGATGCTCTTGACCTTGT | CACCGTGAAATGTCTCTGCT | 55.00 | (CCTTT)5 |
| RYW7 | TCCACTCATCCATTGCTCGT | GATGGATTCAAAGGGACGCT | 55.65 | (CGCGC)5 |
| RYW8 | GGGTCAGAGAATACACAGCG | GTAGGGAAGGAGAAGTGGGT | 55.90 | (AATAG)5 |
| RYW9 | GGACCCTTCCCTCACAGATT | TCCAGTTGCTCTTGCCGTT | 57.10 | (CTAG)6 |
| RYW11 | TGCTCGTCTTCTCGCTTCG | AGTAGTCCTCCACCGCCATCT | 59.20 | (GGTA)5 |
| RYW16 | ATCTCCTCCGCCTTCTAACCC | TGGCAATGGTCGTACAAACT | 56.65 | (GAGC)5 |
| RYW18 | CTCCCTCTTTGTCCTCGTT | GCTGCCTCTTCGCTATCTT | 54.45 | (AGTT)6 |
| RYW19 | GAATGATAGGTCCGCAAGG | CAGCCTTTGTTCAGTTGTCTC | 53.45 | (TTAT)5 |
| RYW28 | CCAAGGCTGAGCAGAAAGAT | ACAAGGTGAAACCCGAAGC | 55.40 | (AGGC)5 |
| RYW31 | ACCCAGAGTCCAGAGAAGC | GATGTCCTCCTCCTTCTCC | 55.60 | (AGCG)5 |
| RYW37 | CATTCCGTTCCTTGTCTTCC | CAGTCTCACTCCTGCGATGT | 55.30 | (GCGAT)5 |
| RYW40 | TGCTCTTCGGCTCTTCTCC | ATCAGCTCATCGTGACCCC | 57.35 | (CAGC)6 |
| RYW49 | GCTAAATCCGCTGATGAGGT | TGTATGTTGCTCCAGCCTTG | 55.05 | (TATC)6 |
| RYW53 | ATGCCTCCGATGTAGATGC | GCCGCCTTCTCTTCATTCT | 54.90 | (GAGG)5 |
| RYW54 | GCACTTGCTCCTGCTTCTC | GACCTTGCCGATGTTGTTG | 55.65 | (CCTC)5 |
| RYW62 | GTTTAGAGAGCAGGAGGCG | AGCCCTGTCCACCCTAATC | 51.40 | (GCTC)5 |
| RYW65 | TAGCGTCGTCAAGAAGCACT | ACAGAGGTGACGAAACGATG | 55.40 | (GCCG)5 |
| RYW67 | GAAGGAAACGCACCAGAGT | TTGGGTTTGTGCTTGGAGT | 55.25 | (TGCG)5 |
| RYW77 | CAGCAAGCAGACAGAGCAG | ACAAAGCAGAGAGGGGAAG | 55.65 | (ATAC)5 |
| RYW84 | AACCCACCCATCCATTCCT | TAAGTCGTCGTCGGCAGAGT | 57.35 | (GAGC)5 |
| Primer name | Na | Ne | I | Ho | He | Nei | PIC |
|---|---|---|---|---|---|---|---|
| RYW5 | 3 | 2.9882 | 1.0967 | 0.7269 | 0.6666 | 0.6654 | 0.7386 |
| RYW7 | 3 | 2.7590 | 1.0507 | 0.7647 | 0.6388 | 0.6375 | 0.6800 |
| RYW8 | 3 | 2.8961 | 1.0806 | 0.7886 | 0.6560 | 0.6547 | 0.6948 |
| RYW9 | 3 | 2.6550 | 1.0260 | 0.7787 | 0.6246 | 0.6233 | 0.6323 |
| RYW11 | 3 | 2.7528 | 1.0508 | 0.6298 | 0.6381 | 0.6367 | 0.7295 |
| RYW16 | 3 | 2.4145 | 0.9578 | 0.5443 | 0.5871 | 0.5858 | 0.6610 |
| RYW18 | 3 | 2.8721 | 1.0771 | 0.6615 | 0.6531 | 0.6518 | 0.7384 |
| RYW19 | 3 | 2.9016 | 1.0808 | 0.7331 | 0.6567 | 0.6554 | 0.6589 |
| RYW28 | 3 | 2.6818 | 1.0364 | 0.6008 | 0.6284 | 0.6271 | 0.7524 |
| RYW31 | 3 | 2.7525 | 1.0499 | 0.7433 | 0.6379 | 0.6367 | 0.6697 |
| RYW37 | 3 | 2.8959 | 1.0797 | 0.7305 | 0.6560 | 0.6547 | 0.7530 |
| RYW40 | 3 | 2.8460 | 1.0698 | 0.7287 | 0.6499 | 0.6486 | 0.6904 |
| RYW49 | 3 | 2.8214 | 1.0646 | 0.7448 | 0.6469 | 0.6456 | 0.7151 |
| RYW53 | 3 | 2.7093 | 1.0417 | 0.6641 | 0.6321 | 0.6309 | 0.7138 |
| RYW54 | 3 | 2.8518 | 1.0708 | 0.7336 | 0.6506 | 0.6493 | 0.7325 |
| RYW62 | 3 | 2.8462 | 1.0699 | 0.7553 | 0.6500 | 0.6487 | 0.6492 |
| RYW65 | 3 | 2.9532 | 1.0904 | 0.7440 | 0.6627 | 0.6614 | 0.6801 |
| RYW67 | 3 | 2.9700 | 1.0935 | 0.7237 | 0.6646 | 0.6633 | 0.6878 |
| RYW77 | 3 | 2.5914 | 1.0094 | 0.7747 | 0.6153 | 0.6141 | 0.6044 |
| RYW84 | 3 | 2.5867 | 1.0066 | 0.7265 | 0.6147 | 0.6134 | 0.6607 |
| Mean | 3 | 2.7873 | 1.0552 | 0.7149 | 0.6415 | 0.6402 | 0.6921 |
| Standard deviation | 0 | 0.1474 | 0.0348 | 0.0632 | 0.0200 | 0.0200 |
表3 20对SSR引物的遗传多样性参数
Table 3 Genetic diversity parameters of 20 pairs of SSR primers
| Primer name | Na | Ne | I | Ho | He | Nei | PIC |
|---|---|---|---|---|---|---|---|
| RYW5 | 3 | 2.9882 | 1.0967 | 0.7269 | 0.6666 | 0.6654 | 0.7386 |
| RYW7 | 3 | 2.7590 | 1.0507 | 0.7647 | 0.6388 | 0.6375 | 0.6800 |
| RYW8 | 3 | 2.8961 | 1.0806 | 0.7886 | 0.6560 | 0.6547 | 0.6948 |
| RYW9 | 3 | 2.6550 | 1.0260 | 0.7787 | 0.6246 | 0.6233 | 0.6323 |
| RYW11 | 3 | 2.7528 | 1.0508 | 0.6298 | 0.6381 | 0.6367 | 0.7295 |
| RYW16 | 3 | 2.4145 | 0.9578 | 0.5443 | 0.5871 | 0.5858 | 0.6610 |
| RYW18 | 3 | 2.8721 | 1.0771 | 0.6615 | 0.6531 | 0.6518 | 0.7384 |
| RYW19 | 3 | 2.9016 | 1.0808 | 0.7331 | 0.6567 | 0.6554 | 0.6589 |
| RYW28 | 3 | 2.6818 | 1.0364 | 0.6008 | 0.6284 | 0.6271 | 0.7524 |
| RYW31 | 3 | 2.7525 | 1.0499 | 0.7433 | 0.6379 | 0.6367 | 0.6697 |
| RYW37 | 3 | 2.8959 | 1.0797 | 0.7305 | 0.6560 | 0.6547 | 0.7530 |
| RYW40 | 3 | 2.8460 | 1.0698 | 0.7287 | 0.6499 | 0.6486 | 0.6904 |
| RYW49 | 3 | 2.8214 | 1.0646 | 0.7448 | 0.6469 | 0.6456 | 0.7151 |
| RYW53 | 3 | 2.7093 | 1.0417 | 0.6641 | 0.6321 | 0.6309 | 0.7138 |
| RYW54 | 3 | 2.8518 | 1.0708 | 0.7336 | 0.6506 | 0.6493 | 0.7325 |
| RYW62 | 3 | 2.8462 | 1.0699 | 0.7553 | 0.6500 | 0.6487 | 0.6492 |
| RYW65 | 3 | 2.9532 | 1.0904 | 0.7440 | 0.6627 | 0.6614 | 0.6801 |
| RYW67 | 3 | 2.9700 | 1.0935 | 0.7237 | 0.6646 | 0.6633 | 0.6878 |
| RYW77 | 3 | 2.5914 | 1.0094 | 0.7747 | 0.6153 | 0.6141 | 0.6044 |
| RYW84 | 3 | 2.5867 | 1.0066 | 0.7265 | 0.6147 | 0.6134 | 0.6607 |
| Mean | 3 | 2.7873 | 1.0552 | 0.7149 | 0.6415 | 0.6402 | 0.6921 |
| Standard deviation | 0 | 0.1474 | 0.0348 | 0.0632 | 0.0200 | 0.0200 |
| Ecotype | Number of sample | Na | Ne | I | Ho | He | PIC |
|---|---|---|---|---|---|---|---|
| NSP | 90 | 3 | 2.7630±0.1736 | 1.0486±0.0461 | 0.7277±0.0695 | 0.6405±0.0251 | 0.6714 |
| LPSS | 182 | 3 | 2.7829±0.1496 | 1.0544±0.0347 | 0.7087±0.0679 | 0.6415±0.0203 | 0.6953 |
表4 不同生态区糜子的遗传多样性参数
Table 4 Genetic diversity parameters of hog millet accessions in different ecotype
| Ecotype | Number of sample | Na | Ne | I | Ho | He | PIC |
|---|---|---|---|---|---|---|---|
| NSP | 90 | 3 | 2.7630±0.1736 | 1.0486±0.0461 | 0.7277±0.0695 | 0.6405±0.0251 | 0.6714 |
| LPSS | 182 | 3 | 2.7829±0.1496 | 1.0544±0.0347 | 0.7087±0.0679 | 0.6415±0.0203 | 0.6953 |
| Origin | Number of sample | Na | Ne | I | Ho | He | PIC |
|---|---|---|---|---|---|---|---|
| Taiyuan | 37 | 2.9545±0.2107 | 2.6940±0.2506 | 1.0247±0.0903 | 0.7306±0.0957 | 0.6344±0.0422 | 0.6353 |
| Datong | 42 | 3.0000±0.0000 | 2.6317±0.1825 | 1.0217±0.0470 | 0.7473±0.0857 | 0.6293±0.0265 | 0.6125 |
| Shuozhou | 17 | 2.9773±0.1508 | 2.6450±0.2514 | 1.0143±0.0840 | 0.7577±0.1181 | 0.6384±0.0415 | 0.5988 |
| Xinzhou | 36 | 3.0000±0.0000 | 2.7119±0.2177 | 1.0335±0.0670 | 0.7258±0.0910 | 0.6382±0.0336 | 0.6423 |
| Lüliang | 17 | 3.0000±0.0000 | 2.5655±0.2810 | 0.9922±0.0851 | 0.7762±0.1155 | 0.6248±0.0452 | 0.5573 |
| Yangquan | 26 | 3.0000±0.0000 | 2.1654±0.2276 | 1.0085±0.0634 | 0.7615±0.1189 | 0.6279±0.0348 | 0.5902 |
| Jinzhong | 18 | 3.0000±0.0000 | 2.6255±0.2668 | 1.0114±0.0752 | 0.7430±0.1297 | 0.6341±0.0431 | 0.6047 |
| Linfen | 11 | 2.9545±0.2107 | 2.6027±0.2994 | 0.9992±0.1018 | 0.7436±0.1383 | 0.6422±0.0525 | 0.5783 |
| Changzhi | 6 | 2.9773±0.1508 | 2.5027±0.3170 | 0.9761±0.0925 | 0.7708±0.1920 | 0.6530±0.0565 | 0.4864 |
| Jincheng | 17 | 2.9773±0.1508 | 2.5844±0.2933 | 0.9967±0.0948 | 0.7234±0.1401 | 0.6286±0.0501 | 0.5976 |
| Yuncheng | 8 | 2.9545±0.2107 | 2.5419±0.3481 | 0.9803±0.1264 | 0.7634±0.1741 | 0.6405±0.0782 | 0.5233 |
表5 不同来源糜子的遗传多样性参数
Table 5 Genetic diversity parameters of hog millet from different source
| Origin | Number of sample | Na | Ne | I | Ho | He | PIC |
|---|---|---|---|---|---|---|---|
| Taiyuan | 37 | 2.9545±0.2107 | 2.6940±0.2506 | 1.0247±0.0903 | 0.7306±0.0957 | 0.6344±0.0422 | 0.6353 |
| Datong | 42 | 3.0000±0.0000 | 2.6317±0.1825 | 1.0217±0.0470 | 0.7473±0.0857 | 0.6293±0.0265 | 0.6125 |
| Shuozhou | 17 | 2.9773±0.1508 | 2.6450±0.2514 | 1.0143±0.0840 | 0.7577±0.1181 | 0.6384±0.0415 | 0.5988 |
| Xinzhou | 36 | 3.0000±0.0000 | 2.7119±0.2177 | 1.0335±0.0670 | 0.7258±0.0910 | 0.6382±0.0336 | 0.6423 |
| Lüliang | 17 | 3.0000±0.0000 | 2.5655±0.2810 | 0.9922±0.0851 | 0.7762±0.1155 | 0.6248±0.0452 | 0.5573 |
| Yangquan | 26 | 3.0000±0.0000 | 2.1654±0.2276 | 1.0085±0.0634 | 0.7615±0.1189 | 0.6279±0.0348 | 0.5902 |
| Jinzhong | 18 | 3.0000±0.0000 | 2.6255±0.2668 | 1.0114±0.0752 | 0.7430±0.1297 | 0.6341±0.0431 | 0.6047 |
| Linfen | 11 | 2.9545±0.2107 | 2.6027±0.2994 | 0.9992±0.1018 | 0.7436±0.1383 | 0.6422±0.0525 | 0.5783 |
| Changzhi | 6 | 2.9773±0.1508 | 2.5027±0.3170 | 0.9761±0.0925 | 0.7708±0.1920 | 0.6530±0.0565 | 0.4864 |
| Jincheng | 17 | 2.9773±0.1508 | 2.5844±0.2933 | 0.9967±0.0948 | 0.7234±0.1401 | 0.6286±0.0501 | 0.5976 |
| Yuncheng | 8 | 2.9545±0.2107 | 2.5419±0.3481 | 0.9803±0.1264 | 0.7634±0.1741 | 0.6405±0.0782 | 0.5233 |
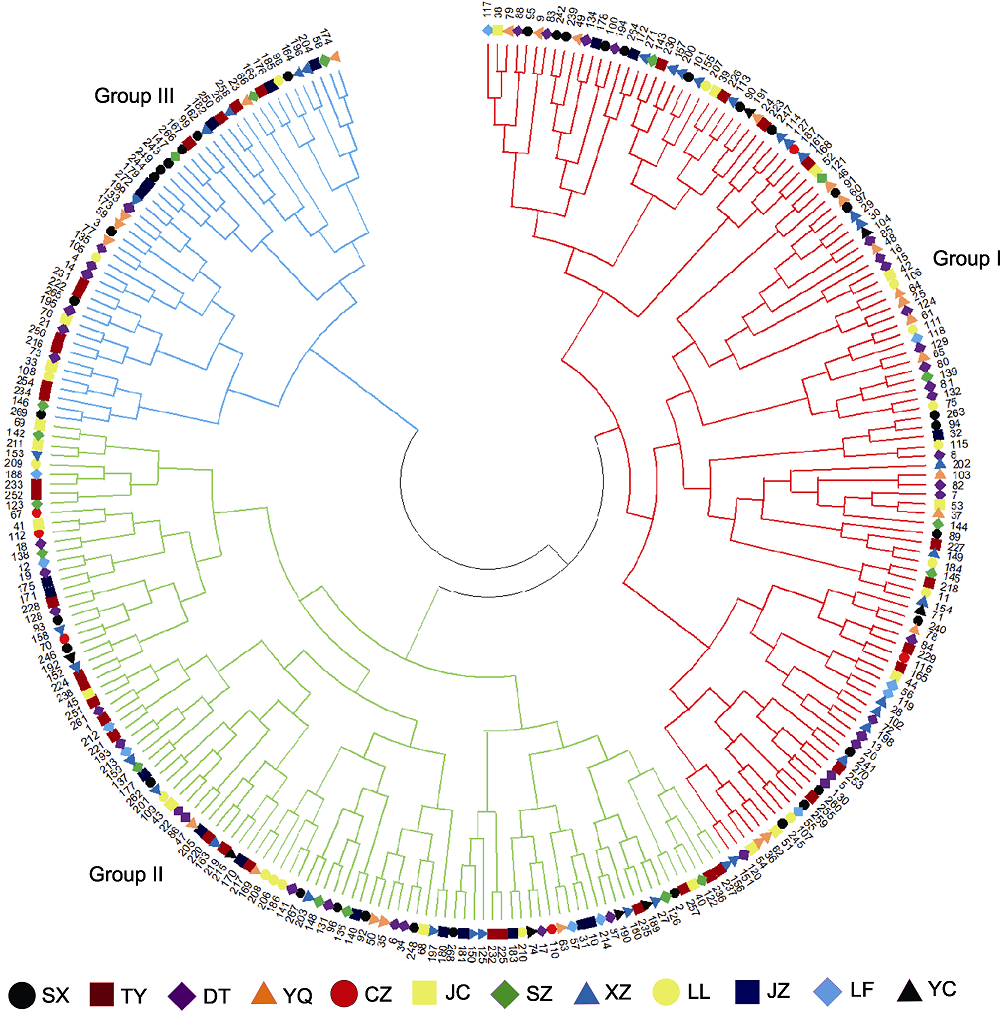
图2 基于遗传距离的272份糜子种质资源聚类 SX: 山西; TY: 太原; DT: 大同; YQ: 阳泉; CZ: 长治; JC: 晋城; SZ: 朔州; XZ: 忻州; LL: 吕梁; JZ: 晋中; LF: 临汾; YC: 运城
Figure 2 Cluster diagram of 272 hog millet accessions based on genetic distance SX: Shanxi; TY: Taiyuan; DT: Datong; YQ: Yangquan; CZ: Changzhi; JC: Jincheng; SZ: Shuozhou; XZ: Xinzhou; LL: Lüliang; JZ: Jinzhong; LF: Linfen; YC: Yuncheng
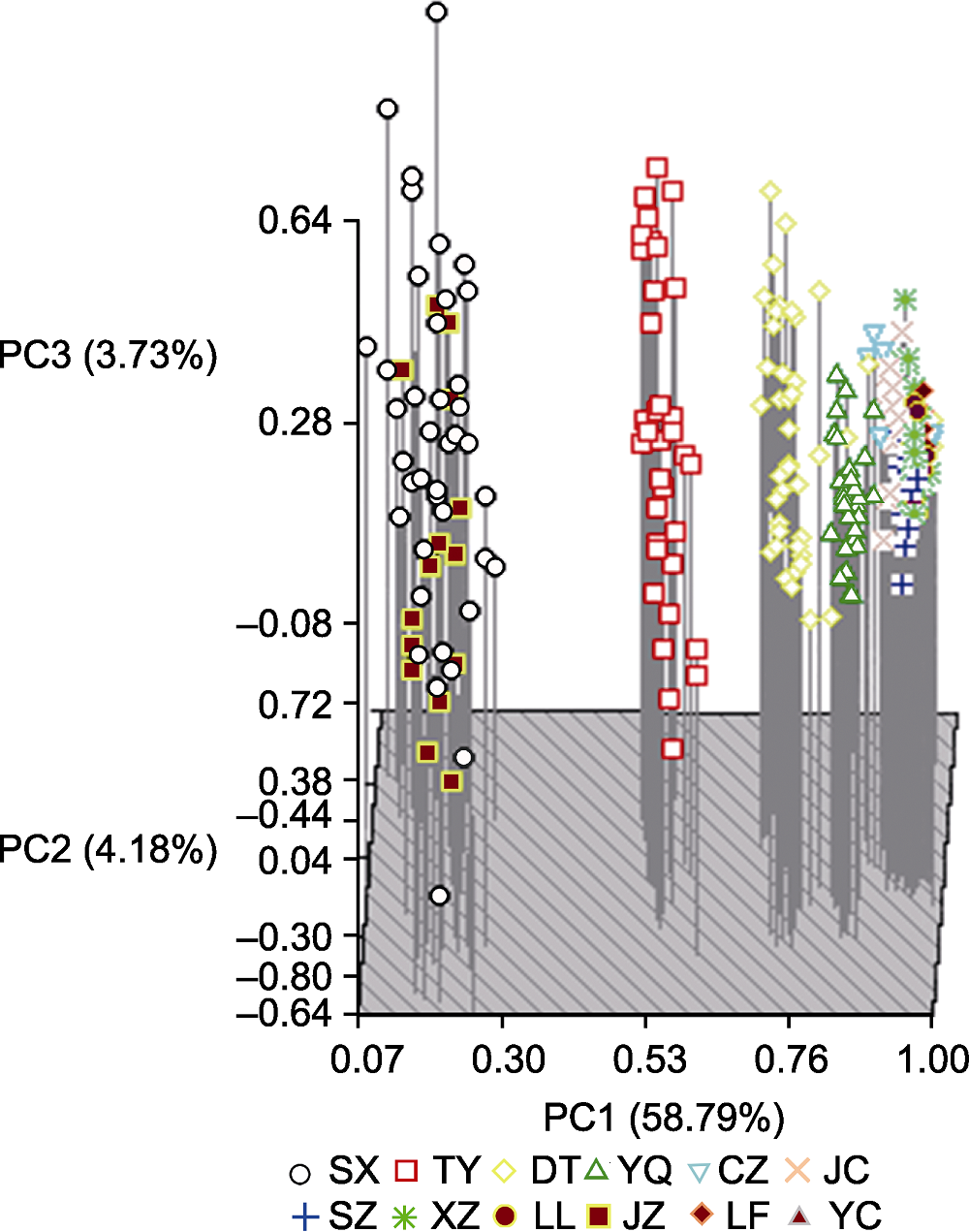
图3 272份糜子材料的主成分分析 SX、TY、DT、YQ、CZ、JC、SZ、XZ、LL、JZ、LF和YC同图2。
Figure 3 Principal component analysis of 272 hog millet materials SX, TY, DT, YQ, CZ, JC, SZ, XZ, LL, JZ, LF, and YC are the same as in Figure 2.
| [1] |
陈昌文, 曹珂, 王力荣, 朱更瑞, 方伟超 (2011). 中国桃主要品种资源及其野生近缘种的分子身份证构建. 中国农业科学 44, 2081-2093.
DOI |
| [2] |
陈小红, 林元香, 王倩, 丁敏, 王海岗, 陈凌, 高志军, 王瑞云, 乔治军 (2022). 基于高基元SSR构建黍稷种质资源的分子身份证. 作物学报 48, 908-919.
DOI |
| [3] |
樊晓静, 于文涛, 蔡春平, 林浥, 王泽涵, 房婉萍, 张见明, 叶乃兴 (2021). 利用SNP标记构建茶树品种资源分子身份证. 中国农业科学 54, 1751-1760.
DOI |
| [4] |
高源, 刘凤之, 王昆, 王大江, 龚欣, 刘立军 (2015). 苹果部分种质资源分子身份证的构建. 中国农业科学 48, 3887-3898.
DOI |
| [5] | 管俊娇, 杨龙, 木万福, 杨晓洪, 张鹏, 黄清梅, 张建华 (2021). 利用SSR标记构建青花菜的分子身份证. 中国蔬菜 (12), 39-45. |
| [6] |
郭琪, 郭大龙, 郭丽丽, 张琳, 侯小改 (2015). SSR分子标记在牡丹亲缘关系研究中的应用与研究进展. 植物学报 50, 652-664.
DOI |
| [7] |
何杰丽, 石甜甜, 陈凌, 王海岗, 高志军, 杨美红, 王瑞云, 乔治军 (2019). 糜子EST-SSR分子标记的开发及种质资源遗传多样性分析. 植物学报 54, 723-732.
DOI |
| [8] | 侯丽媛, 张春芬, 邓舒, 肖蓉, 赵菁, 孟玉平, 曹秋芬 (2020). 苹果新品种赤霞和栽培品种遗传多样性分析和分子身份证构建. 分子植物育种 18, 7588-7599. |
| [9] | 胡振帮, 高运来, 齐照明, 蒋洪蔚, 刘春燕, 辛大伟, 胡国华, 潘校成, 陈庆山 (2016). 作物分子身份证构建软件ID analysis的编制. 中国农业科学 49, 2255-2266. |
| [10] | 李法鹏 (2019). 花生种质资源遗传多样性研究. 硕士论文. 郑州: 河南农业大学. pp. 1-3. |
| [11] | 李红琴, 刘宝龙, 张波, 张怀刚 (2020). 青海省审定小麦品种SSR遗传多样性分析及分子身份证的建立. 作物杂志 (3), 60-65. |
| [12] |
连帅, 陆平, 乔治军, 张琦, 张茜, 刘敏轩, 王瑞云 (2016). 利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性. 中国农业科学 49, 3264-3275.
DOI |
| [13] | 林元香, 陈小红, 王蓉, 石甜甜, 陈凌, 王海岗, 乔治军, 王瑞云 (2020). 中国和印度黍稷的遗传差异分析. 山西农业科学 48, 865-871. |
| [14] | 刘敏轩, 许月, 陆平 (2020). 中国野生黍稷资源收集保存与遗传多样性研究进展. 植物遗传资源学报 21, 1435-1445. |
| [15] |
陆徐忠, 倪金龙, 李莉, 汪秀峰, 马卉, 张小娟, 杨剑波 (2014). 利用SSR分子指纹和商品信息构建水稻品种身份证. 作物学报 40, 823-829.
DOI |
| [16] | 马琳, 刘海珍, 陆徐忠, 倪金龙, 张小娟, 杨剑波 (2013). 130份甘蓝型油菜种质分子身份证的构建. 中国油料作物学报 35, 231-239. |
| [17] | 倪西源, 柳寒, 黄吉祥, 石江华, 赵坚义 (2020). 利用InDel标记构建甘蓝型油菜的分子身份证. 分子植物育种 18, 4671-4679. |
| [18] | 冉昆, 隋静, 王宏伟, 魏树伟, 张勇, 董冉, 董肖昌, 王少敏 (2018). 利用SSR荧光标记构建山东地方梨种质资源分子身份证. 果树学报 35(S1), 71-78. |
| [19] |
石甜甜, 何杰丽, 高志军, 陈凌, 王海岗, 乔治军, 王瑞云 (2019). 利用EST-SSR评估糜子资源遗传差异. 中国农业科学 52, 4100-4109.
DOI |
| [20] | 陶杰, 张江江, 常丽, 张翠萍, 李建军, 张超, 李德芳, 赵立宁 (2022). 盐胁迫下大麻SSR标记的分布特征分析. 中国麻业科学 44, 5-12. |
| [21] | 王凤格, 杨扬, 易红梅, 赵久然, 任洁, 王璐, 葛建镕, 江彬, 张宪晨, 田红丽, 侯振华 (2017). 中国玉米审定品种标准SSR指纹库的构建. 中国农业科学 50, 1-14. |
| [22] | 王纶, 王星玉, 温琪汾, 武变娥 (2005). 中国黍稷种质资源研究与利用. 植物遗传资源学报 6, 474-477. |
| [23] |
王瑞云, 刘笑瑜, 王海岗, 陆平, 刘敏轩, 陈凌, 乔治军 (2017). 用高基元微卫星标记分析中国糜子遗传多样性. 中国农业科学 50, 3848-3859.
DOI |
| [24] | 王舒婷, 何杰丽, 石甜甜, 陈凌, 王海岗, 王瑞云, 乔治军 (2019). 利用微卫星标记分析山西糜子的遗传多样性. 植物遗传资源学报 20, 69-78. |
| [25] | 王星玉, 王纶, 温琪汾, 师颖 (2009). 山西是黍稷的起源和遗传多样性中心. 植物遗传资源学报 10, 465-470, 474. |
| [26] |
咸丰, 路战远, 张建中, 陈立宇, 程玉臣, 杨建强, 张向前, 苏和 (2022). 基于SSR分子标记的早熟棉种质资源遗传多样性分析. 北方农业学报 50, 11-17.
DOI |
| [27] |
薛延桃, 陆平, 乔治军, 刘敏轩, 王瑞云 (2018). 基于SSR标记的黍稷种质资源遗传多样性及亲缘关系研究. 中国农业科学 51, 2846-2859.
DOI |
| [28] |
杨文娟, 张艳欣, 王林海, 魏鑫, 黎冬华, 高媛, 刘盼, 张秀荣 (2018). 一个芝麻应用核心种质的DNA分子身份证构建. 作物学报 44, 1010-1026.
DOI |
| [29] | 赵久然, 王凤格, 郭景伦, 陈刚, 廖琴, 孙世贤, 陈如明, 刘龙洲 (2003). 中国玉米新品种DNA指纹库建立系列研究II. 适于玉米自交系和杂交种指纹图谱绘制的SSR核心引物的确定. 玉米科学 11(2), 3-5, 8. |
| [30] | 赵淑菊, 王丽娜 (2005). 糜子品种引种试验初报. 杂粮作物 25, 180-181. |
| [31] |
左茜茜, 宋英杰, 马心妍, 杨云卉, 王轶菲, 郭泽光, 朱雄智, 刘越 (2022). 苦荞全基因组SSR位点挖掘及遗传多样性分析. 中国农业科技导报 24, 38-51.
DOI |
| [32] |
Amadou I, Gounga ME, Le GW (2013). Millets: nutritional composition, some health benefits and processing—a review. Emir J Food Agric 25, 501-508.
DOI URL |
| [33] |
Barton L, Newsome SD, Chen FH, Wang H, Guilderson TP, Bettinger RL (2009). Agricultural origins and the isotopic identity of domestication in northern China. Proc Natl Acad Sci USA 106, 5523-5528.
DOI PMID |
| [34] |
Cao XN, Hu YL, Song J, Feng H, Wang JJ, Chen L, Wang L, Diao XM, Wan Y, Liu SC, Qiao ZJ (2022). Transcriptome sequencing and metabolome analysis reveals the molecular mechanism of drought stress in millet. Int J Mol Sci 23, 10792.
DOI URL |
| [35] |
Crawford GW (2009). Agricultural origins in North China pushed back to the Pleistocene-Holocene boundary. Proc Natl Acad Sci USA 106, 7271-7272.
DOI PMID |
| [36] |
Hu XY, Wang JF, Lu P, Zhang HS (2009). Assessment of genetic diversity in broomcorn millet (Panicum miliaceum L.) using SSR markers. J Genet Genomics 36, 491-500.
DOI URL |
| [37] |
Hunt HV, Rudzinski A, Jiang HE, Wang RY, Thomas MG, Jones MK (2018). Genetic evidence for a western Chinese origin of broomcorn millet (Panicum miliaceum). Holocene 28, 1968-1978.
DOI URL |
| [38] |
Liu KJ, Muse SV (2005). PowerMarker: an integrated analysis environment for genetic marker analysis. Bioinformatics 21, 2128-2129.
DOI PMID |
| [39] |
Murray MG, Thompson WF (1980). Rapid isolation of high molecular weight plant DNA. Nucleic Acids Res 8, 4321-4326.
DOI PMID |
| [40] |
Na XF, Cao XN, Ma CX, Ma SL, Xu PX, Liu SC, Wang JJ, Wang HG, Chen L, Qiao ZJ (2019). Plant stage, not drought stress, determines the effect of cultivars on bacterial community diversity in the rhizosphere of broomcorn millet (Panicum miliaceum L.). Front Microbiol 10, 828.
DOI URL |
| [41] |
Saha D, Gowda MVC, Arya L, Verma M, Bansal KC (2016). Genetic and genomic resources of small millets. Cri Rev Plant Sci 35, 56-79.
DOI URL |
| [42] |
Saleh ASM, Zhang Q, Chen J, Shen Q (2013). Millet grains: nutritional quality, processing, and potential health benefits. Compr Rev Food Sci Food Saf 12, 281-295.
DOI URL |
| [43] |
Song YS, Suwabe K, Wako T, Ohara T, Nunome T, Kojima A (2004). Development of microsatellite markers in bunching onion (Allium fistulosum L.). Breed Sci 54, 361-365.
DOI URL |
| [44] |
Tóth G, Gáspári Z, Jurka J (2000). Microsatellites in different eukaryotic genomes: survey and analysis. Genome Res 10, 967-981.
DOI PMID |
| [45] |
Tsukazaki H, Yamashita K, Yaguchi S, Masuzaki S, Fukuoka H, Yonemaru J, Kanamori H, Kono I, Hang TTM, Shigyo M, Kojima A, Wako T (2008). Construction of SSR-based chromosome map in bunching onion (Allium fistulosum). Theor Appl Genet 117, 1213-1223.
DOI PMID |
| [46] |
Varshney RK, Graner A, Sorrells ME (2005). Genic microsatellite markers in plants: features and applications. Trends Biotechnol 23, 48-55.
DOI PMID |
| [47] |
Ventura F, Vignudelli M, Poggi GM, Negri L, Dinelli G (2020). Phenological stages of Proso millet (Panicum miliaceum L.) encoded in BBCH scale. Int J Biometeorol 64, 1167-1181.
DOI |
| [48] |
Wang BH, Zhu P, Yuan YL, Wang CB, Yu CM, Zhang HH, Zhu XY, Wang W, Yao CB, Zhuang ZM, Li P (2014). Development of EST-SSR markers related to salt tolerance and their application in genetic diversity and evolution analysis in Gossypium. Genet Mol Res 13, 3732-3746.
DOI PMID |
| [49] |
Webb A, Knoblauch J, Sabankar N, Kallur AS, Hey J, Sethuraman A (2021). The pop-gen pipeline platform: a software platform for population genomic analyses. Mol Biol Evol 38, 3478-3485.
DOI URL |
| [1] | 段政勇, 丁敏, 王宇卓, 丁艺冰, 陈凌, 王瑞云, 乔治军. 糜子SBP基因家族全基因组鉴定及表达分析[J]. 植物学报, 2024, 59(2): 231-244. |
| [2] | 杨清华, 王洪露, 冯佰利. 糜子品质研究进展与展望[J]. 植物学报, 2023, 58(1): 22-33. |
| [3] | 刘佳佳, 张大众, 张渊博, 张楚楚, 周佳玥, 熊亚红, 卓振生, 饶煜健, 冯佰利. 硒和碲对缓解糜子镉毒害及减少籽粒镉积累的调控效应[J]. 植物学报, 2023, 58(1): 62-76. |
| [4] | 陈向向, 盖中帅, 翟军团, 徐劲东, 焦培培, 吴智华, 李志军. 中国西北地区天然胡杨群体遗传多样性及核心保护单元的构建[J]. 生物多样性, 2021, 29(12): 1638-1649. |
| [5] | 吴漫玲, 姚兰, 艾训儒, 朱江, 朱强, 王进, 黄小, 洪建峰. 水杉原生种群核心种质资源的繁殖特性[J]. 生物多样性, 2020, 28(3): 303-313. |
| [6] | 何杰丽,石甜甜,陈凌,王海岗,高志军,杨美红,王瑞云,乔治军. 糜子EST-SSR分子标记的开发及种质资源遗传多样性分析[J]. 植物学报, 2019, 54(6): 723-732. |
| [7] | 薛建华, 姜莉, 马晓林, 邴艳红, 赵思晨, 马克平. 莲品种DNA指纹图谱的构建[J]. 生物多样性, 2016, 24(1): 3-11. |
| [8] | 郭数进,李玮瑜,马艳芸,赵恒,乔玲,李贵全. 山西不同生态型大豆品种苗期耐低温性综合评价[J]. 植物生态学报, 2014, 38(9): 990-1000. |
| [9] | 张洋, 谭敦炎. 地下结实植物白番红花的繁育系统与传粉生物学[J]. 生物多样性, 2009, 17(5): 468-475. |
| [10] | 李长涛 宋丽丽 孔伟丽 洪兆龙. 植物遗传资源核心种质的构建及应用[J]. 植物学报, 2005, 22(增刊): 139-145. |
| [11] | 吐米尔, 阿不都拉·阿巴斯, 热衣木江·马木提, 孜比尔尼沙·吾买尔. 新疆天山西部山脉森林生态系统地衣群落结构的初步研究[J]. 植物生态学报, 2003, 27(6): 810-816. |
| [12] | 张国钢, 张正旺, 郑光美, 李晓强, 李俊峰, 黄雷. 山西五鹿山褐马鸡不同季节的空间分布与栖息地选择研究[J]. 生物多样性, 2003, 11(4): 303-308. |
| [13] | 张峰, 上官铁梁. 山西翅果油树群落优势种群分布格局研究[J]. 植物生态学报, 2000, 24(5): 590-594. |
| [14] | 米湘成, 张金屯, 张峰, 上官铁梁, 李爱华, 郑凤英. 山西高原植被与土壤分布格局关系的研究[J]. 植物生态学报, 1999, 23(4): 336-344. |
| [15] | 米湘成, 张金屯, 张峰, 上官铁梁, 郑凤英. 山西高原植被与气候的关系分析及植被数量区划的研究[J]. 植物生态学报, 1996, 20(6): 549-560. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||