

植物学报 ›› 2020, Vol. 55 ›› Issue (1): 1-4.DOI: 10.11983/CBB19240 cstr: 32102.14.CBB19240
• 热点评 • 下一篇
收稿日期:2019-12-15
接受日期:2019-12-17
出版日期:2020-01-01
发布日期:2019-12-20
通讯作者:
黄学辉
基金资助:
Jiali Tang,Jie Qiu,Xuehui Huang( )
)
Received:2019-12-15
Accepted:2019-12-17
Online:2020-01-01
Published:2019-12-20
Contact:
Xuehui Huang
摘要: 园艺植物包括花卉、蔬菜、果树、部分瓜类(如西瓜(Citrullus lanatus)和甜瓜(Cucumis melo))和茶树(Camellia sinensis), 在植物分类上涉及大量物种。园艺植物的基因组学和遗传学研究具有重要的理论价值和经济意义。基因组测序技术及相关生物信息学工具的发展为园艺植物基因组和分子生物学研究注入了新的活力。睡莲是一种重要的花卉植物, 除了具有观赏价值, 其进化地位也非常特殊, 属于一种早期被子植物类群。最近, 蓝星睡莲(N. colorata)的高质量基因组图谱绘制完成。通过系统分析和比较睡莲基因组与其它被子植物的基因组, 研究者阐明了睡莲的进化位置及相关进化事件。所获得的高质量基因组序列将有助于园艺植物研究者开展深入的分子遗传学研究, 鉴定到控制和调控花器官、花色花香及品质等众多性状的功能基因, 从而推动基础研究的快速发展和加快新品种创制。
唐嘉瓅,邱杰,黄学辉. 基因组学技术大发展助力园艺植物研究取得新进展. 植物学报, 2020, 55(1): 1-4.
Jiali Tang,Jie Qiu,Xuehui Huang. The Development of Genomics Technologies Drives New Progress in Horticultural Plant Research. Chinese Bulletin of Botany, 2020, 55(1): 1-4.
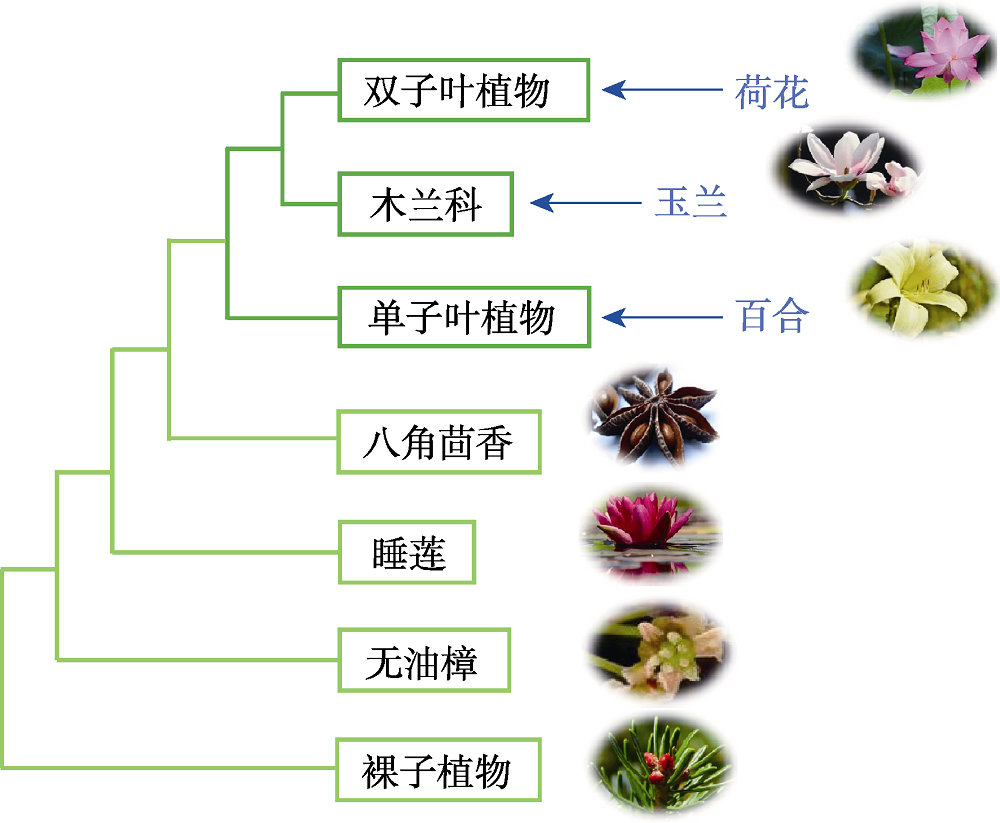
图1 植物系统发育关系简图 在植物系统发育过程中, 睡莲是比较基础的原始被子植物, 而荷花(Nelumbo nucifera)位于高等的双子叶植物中。
Figure 1 Plant phylogeny diagram According to plant phylogeny, water lilies belong to primitive angiosperms, while lotuses belong to typical dicotyledons.
| [1] | Chen F, Liu X, Yu CW, Chen YC, Tang HB, Zhang LS (2017). Water lilies as emerging models for Darwin’s abominable mystery. Hortic Res 4, 17051. |
| [2] | Chen LY, VanBuren R, Paris M, Zhou HY, Zhang XT, Wai CM, Yan HS, Chen S, Alonge M, Ramakrishnan S, Liao ZY, Liu J, Lin JS, Yue JJ, Fatima M, Lin ZC, Zhang JS, Huang LX, Wang H, Hwa TY, Kao SM, Choi AEY, Sharma A, Song J, Wang LL, Yim WC, Cushman JC, Paull RE, Matsumoto T, Qin Y, Wu QS, Wang JP, Yu QY, Wu J, Zhang SL, Boches P, Tung CW, Wang ML, D’Eeckenbrugge GC, Sanewski GM, Purugganan MD, Schatz MC, Bennetzen JL, Lexer C, Ming R (2019). The bracteatus pineapple genome and domestication of clonally propagated crops. Nat Genet 51, 1549-1558. |
| [3] | Guo SG, Zhao SJ, Sun HH, Wang X, Wu S, Lin T, Ren Y, Gao L, Deng Y, Zhang J, Lu XQ, Zhang HY, Shang JL, Gong GY, Wen CL, He N, Tian SW, Li MY, Liu JP, Wang YP, Zhu YC, Jarrets R, Levi A, Zhang XP, Huang SW, Fei ZJ, Liu WG, Xu Y (2019). Resequencing of 414 cultivated and wild watermelon accessions identifies selection for fruit quality traits. Nat Genet 51, 1616-1623. |
| [4] | Liao NQ, Hu ZY, Li YY, Hao JF, Chen SN, Xue Q, Ma YY, Zhang KJ, Mahmoud A, Ali A, Malangisha GK, Lyu XL, Yang JH, Zhang MF (2020). Ethylene-responsive factor 4 is associated with the desirable rind hardness trait conferring cracking resistance in fresh fruits of watermelon. Plant Biol J 18, 1066-1077. |
| [5] | Ming R, VanBuren R, Wai CM, Tang HB, Schatz MC, Bowers JE, Lyons E, Wang ML, Chen J, Biggers E, Zhang JS, Huang LX, Zhang LM, Miao WJ, Zhang J, Ye ZY, Miao CY, Lin ZC, Wang H, Zhou HY, Yim WC, Priest HD, Zheng CF, Woodhouse M, Edger PP, Guyot R, Guo HB, Guo H, Zheng GY, Singh R, Sharma A, Min XJ, Zheng Y, Lee H, Gurtowski J, Sedlazeck FJ, Harkess A, McKain MR, Liao ZY, Fang JP, Liu J, Zhang XD, Zhang Q, Hu WC, Qin Y, Wang K, Chen LY, Shirley N, Lin YR, Liu LY, Hernandez AG, Wright CL, Bulone V, Tuskan GA, Heath K, Zee F, Moore PH, Sunkar R, Leebens-Mack JH, Mockler T, Bennetzen JL, Freeling M, Sankoff D, Paterson AH, Zhu XG, Yang XH, Smith JAC, Cushman JC, Paull RE, Yu QY (2015). The pineapple genome and the evolution of CAM photosynthesis. Nat Genet 47, 1435-1442. |
| [6] | The French-Italian Public Consortium for Grapevine Genome Characterization (2007). The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature 449, 463-467. |
| [7] | Zhang LS, Chen F, Zhang XT, Li Z, Zhao YY, Rolf L, Chang XJ, Dong W, Simon YWH, Liu X, Song AX, Chen JH, Guo WL, Wang ZJ, Zhuang YY, Wang HF, Chen XQ, Hu J, Liu YH, Qin Y, Wang K, Dong SS, Liu Y, Zhang SZ, Yu XX, Wu Q, Wang LS, Yan XQ, Jiao YN, Kong HZ, Zhou XF, Yu CW, Chen YC, Li F, Wang JH, Chen W, Chen XL, Jia QD, Zhang C, Jiang YF, Zhang WB, Liu GH, Fu JY, Chen F, Ma H, Yves VP, Tang HB (2019). The water lily genome and the early evolution of flowering plants. Nature 577, 79-84. |
| [8] | Zhao GW, Lian Q, Zhang ZH, Fu QS, He YH, Ma S, Ruggieri V, Monforte AJ, Wang PY, Julca I, Wang HS, Liu JP, Xu Y, Wang RZ, Ji JB, Xu ZH, Kong WH, Zhong Y, Shang JL, Pereira L, Argyris J, Zhang J, Mayobre C, Pujol M, Oren E, Out D, Wang JM, Sun DX, Zhao SJ, Zhu YC, Li N, Katzir N, Gur A, Dogimont C, Schaefer H, Fan W, Bendahmane A, Fei Z, Pitrat M, Gabaldon T, Lin T, Garcia-Mas J, Xu YY, Huang SW (2019). A comprehensive genome variation map of melon identifies multiple domestication events and loci influencing agronomic traits. Nat Genet 51, 1607-1615. |
| [1] | 逯子佳, 王天瑞, 郑斯斯, 孟宏虎, 曹建国, Gregor Kozlowski, 宋以刚. 孑遗植物湖北枫杨的环境适应性遗传变异与遗传脆弱性[J]. , 2025, 49(濒危植物的保护与恢复): 0-. |
| [2] | 周鑫宇, 刘会良, 高贝, 卢妤婷, 陶玲庆, 文晓虎, 张岚, 张元明. 新疆特有濒危植物雪白睡莲繁殖生物学研究[J]. , 2025, 49(濒危植物的保护与恢复): 0-. |
| [3] | 卢晓强, 董姗姗, 马月, 徐徐, 邱凤, 臧明月, 万雅琼, 李孪鑫, 于赐刚, 刘燕. 前沿技术在生物多样性研究中的应用现状、挑战与展望[J]. 生物多样性, 2025, 33(4): 24440-. |
| [4] | 王传永, 庄典, 宋正达, 翟恒华, 李乃伟, 张凡. 黑果腺肋花楸叶绿体全基因组的结构和比较分析及系统进化推断[J]. 植物学报, 2025, 60(4): 1-0. |
| [5] | 崔娟, 于晓玉, 于跃娇, 梁铖玮, 孙健, 陈温福. 影响中国东北和日本粳稻食味品质差异的质构因素及其遗传基础解析[J]. 植物学报, 2025, 60(4): 1-0. |
| [6] | 张如礼, 李德铢, 张玉霄. 短穗竹居群遗传结构及气候适应性分析[J]. 植物学报, 2025, 60(3): 407-424. |
| [7] | 林珍, 向家宝, 蔡何佳奕, 高贝, 杨金涛, 李俊毅, 周青松, 黄晓磊, 邓鋆. 七种半翅目昆虫线粒体基因组数据[J]. 生物多样性, 2025, 33(2): 24434-. |
| [8] | 曹东, 李焕龙, 彭扬, 魏存争. 植物基因组大小与性状关系的研究进展[J]. 生物多样性, 2025, 33(2): 24192-. |
| [9] | 夏琳凤, 李瑞, 王海政, 冯大领, 王春阳. 轮藻门植物基因组学研究进展[J]. 植物学报, 2025, 60(2): 271-282. |
| [10] | 邓洪, 钟占友, 寇春妮, 朱书礼, 李跃飞, 夏雨果, 武智, 李捷, 陈蔚涛. 基于线粒体全基因组揭示斑鳠的种群遗传结构与演化历史[J]. 生物多样性, 2025, 33(1): 24241-. |
| [11] | 姚祥坦, 张心怡, 陈阳, 袁晔, 程旺大, 王天瑞, 邱英雄. 基于基因组重测序揭示栽培欧菱遗传多样性及‘南湖菱’的起源驯化历史[J]. 生物多样性, 2024, 32(9): 24212-. |
| [12] | 陈楠, 张全国. 实验进化研究途径[J]. 生物多样性, 2024, 32(9): 24171-. |
| [13] | 李园, 范开建, 安泰, 李聪, 蒋俊霞, 牛皓, 曾伟伟, 衡燕芳, 李虎, 付俊杰, 李慧慧, 黎亮. 玉米自然群体自交系农艺性状的多环境全基因组预测初探[J]. 植物学报, 2024, 59(6): 1041-1053. |
| [14] | 何花, 谭敦炎, 杨晓琛. 被子植物隐性雌雄异株性系统的多样性、系统演化及进化意义[J]. 生物多样性, 2024, 32(6): 24149-. |
| [15] | 张强, 赵振宇, 李平华. 基因编辑技术在玉米中的研究进展[J]. 植物学报, 2024, 59(6): 978-998. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||