
|
|
||
|
基因编辑技术在玉米中的研究进展
植物学报
2024, 59 (6):
978-998.
DOI: 10.11983/CBB24080
基因编辑技术已成为现代农业育种领域的重要工具。玉米(Zea mays)是全球最重要的粮食作物之一, 基因编辑技术在玉米中的应用显示出巨大的潜力。该文综述了基因编辑技术在玉米研究中的应用进展, 重点介绍了CRISPR/Cas等系统在玉米基因组编辑中的最新成果。首先, 介绍了基因编辑技术的基本原理和类型, 特别是CRISPR/Cas系统的工作机制及其在玉米中的应用优势。其次, 总结了基因编辑技术在玉米育种中的研究进展, 涵盖从基础的基因组编辑到复杂的多基因编辑, 旨在改良玉米的产量、品质和抗逆性等关键性状。最后, 文章列举了我国在玉米基因编辑方面的杰出工作, 并讨论了基因编辑技术应用于玉米育种中存在的问题, 同时展望了未来发展方向。 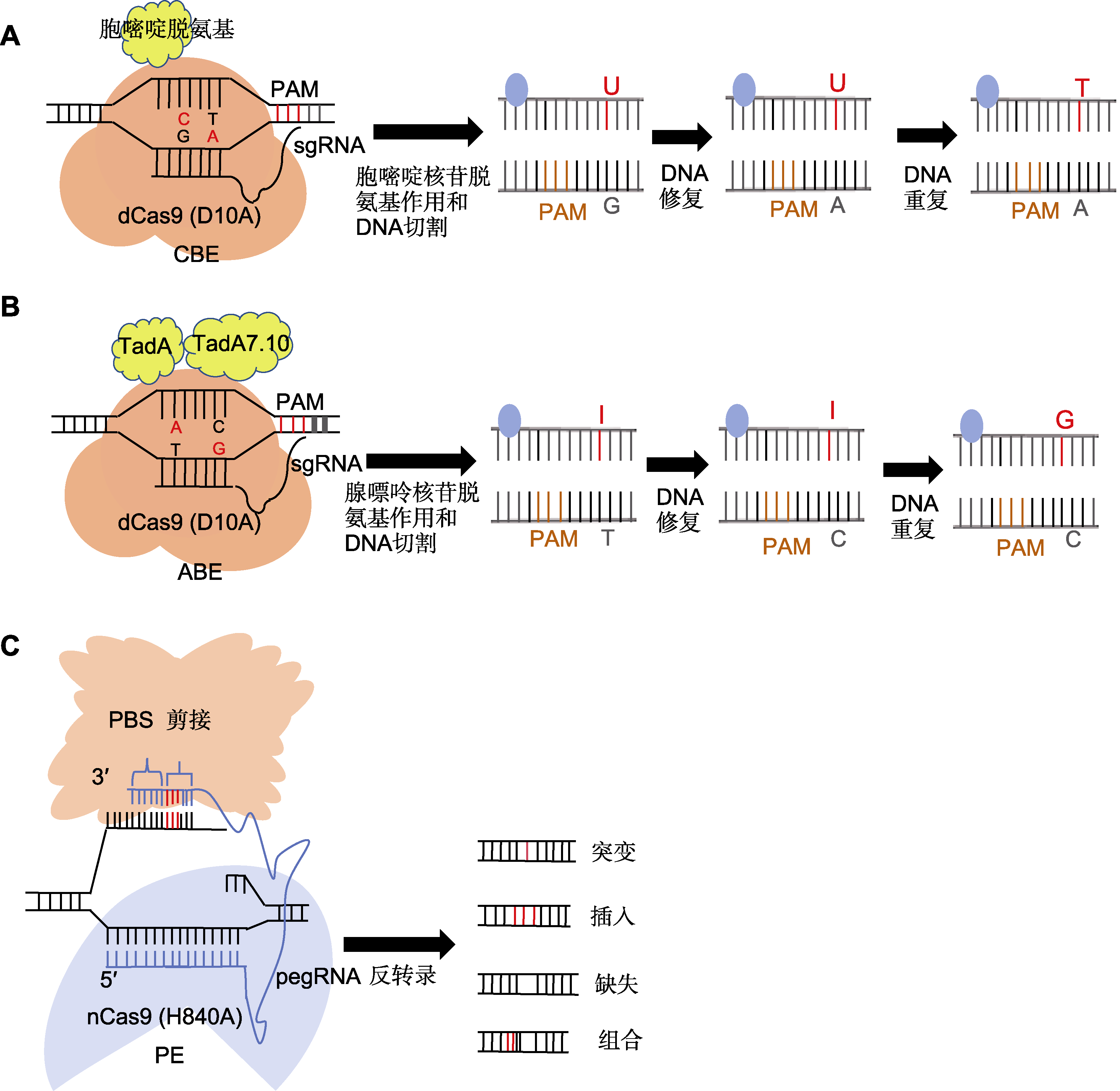 View image in article
图2
碱基编辑器技术工作原理
(A) sgRNA识别并结合到目标DNA序列特定的原间隔区邻近基序(PAM)位点, dCas9蛋白则与sgRNA结合, 形成复合物并结合于目标DNA上, 但不切割DNA双链, 仅使其单链化; 随后胞嘧啶脱氨酶在sgRNA的引导下, 接触到暴露的单链DNA上的胞嘧啶, 并催化其脱氨反应, 将胞嘧啶转化为尿嘧啶; 最后在DNA重复或修复过程中, 尿嘧啶被视为胸腺嘧啶的类似物, 从而纳入到新合成的DNA链中, 实现C-G碱基对到T-A碱基对的直接替换; (B) 利用CRISPR-Cas9系统中的sgRNA识别并结合到目标DNA序列的PAM位点, Cas9蛋白与sgRNA结合形成复合物, 并定位于目标DNA上; ABE中的腺嘌呤脱氨酶在sgRNA的引导下, 接触到暴露的单链DNA上的腺嘌呤, 并催化其脱氨反应, 将腺嘌呤转化为次黄嘌呤或脱氧次黄嘌呤; 最后在DNA修复过程中, 识别到次黄嘌呤后, 启动修复过程, 中间产物通常被替换为鸟嘌呤, 从而实现A-T碱基对到G-C碱基对的直接替换; (C) 利用pegRNA作为引导分子结合sgRNA, 并在其3'末端增加引物结合位点(PBS)序列和逆转录模板(RTT); 在pegRNA的引导下, 部分失活的Cas9切口酶切断含PAM序列的DNA单链; 切割后的DNA单链与pegRNA的3'末端PBS序列互补并结合, 随后逆转录酶沿RTT模板序列开始逆转录反应, 将目标编辑序列直接引到DNA切口处; 随后细胞内DNA修复机制识别并处理切口处的DNA结构, 最终保留携带目标编辑的DNA链。CBE、ABE和PE同
正文中引用本图/表的段落
2016年, David Liu团队在遗传编辑技术上取得了突破性进展, 他们以CRISPR/Cas9系统为基础, 开发出一种新的基因编辑技术——碱基编辑。该系统将胞嘧啶脱氨酶与nCas9或dCas9以及sgRNA结合。该复合体应用于特定的DNA位点, 结合后胞嘧啶脱氨酶被激活, 催化目标DNA链上的胞嘧啶(C)转变成尿嘧啶(U)。这一关键改变影响后续在DNA重复或修复阶段传递, 最终在DNA序列中实现精确的C→T或G→A配对转化(图2A) (Komor et al., 2017)。第一代胞嘧啶碱基编辑系统(cytosine base editor 1, CBE1)由APOBEC1胞嘧啶脱氨酶和CRISPR-Cas9的核酸酶变体(nCas9或dCas9)组成, 并通过一个16氨基酸的XTEN连接成肽, 实现二者的融合。该系统通过避免依赖DSB的设计, 显著降低了基因编辑过程中非目标效应和基因组不稳定性; 并能够将C直接转变为U, 进而在DNA重复或修复过程中被解读为胸腺嘧啶(T), 从而实现C→T的精确点突变。但CBE1在体内的编辑效率较低, 归因于尿嘧啶DNA糖苷酶(UDG)的作用, 该酶通过碱基切除修复(BER)途径识别并移除U-G错配, 修复CBE1进行的C→U编辑。为克服这一限制, 引入了第二代碱基编辑器CBE2 (RAPOBEC1- XTEN-dCas9-UGI), 其中包括一个来自噬菌体PBS的尿嘧啶DNA糖苷酶抑制剂(UGI)。UGI的加入可抑制UDG的活性, 从而提高体内基因编辑效率。与BE1相比, BE2的体内编辑活力明显提高。在CBE1和CBE2的基础上, 第三代碱基编辑器CBE3 (RAPOBEC1-XTEN-nCas9-UGI)采用更精细的策略, 包括一个能进行单链刻痕的活性nCas9 (D10A)变体。这种战略性修改促进了DNA链断裂, 显著增强C→T转换效率, 同时观察到插入或缺失(indels)率约为1.1% (Komor et al., 2016)。相较于CBE3, CBE4有2个关键的改进: 双份UGI的引入和连接APOBEC1与nCas9的扩展连接肽。这些优化措施显著提高了系统编辑效率, 同时最大限度地减少indels和靶外效应(Lee et al., 2020)。
2016年, 引入了一种具有革命性的遗传编辑工具腺嘌呤碱基编辑(adenine base editor, ABE) (Nishida et al., 2016)。该系统与CBE有所不同, 它能够将A-T碱基对直接转化为G-C。ABE系统由携带D10A突变的nCas9和腺苷脱氨酶组成, 在gRNA的精确引导下, ABE能够准确地识别靶向目标DNA序列, 将腺嘌呤(A)转化为腺苷(I)。随后, 在DNA重复过程中, I被解读为鸟嘌呤(G), 从而显著提高基因编辑的精确性(图2B) (Kim et al., 2017)。为促进DNA中腺嘌呤直接脱氨, 研究人员重新定位大肠杆菌tRNA腺苷脱氨酶(ec- TadA), 并通过筛选与定向进化, 成功开发出一种ecTadA突变体, 用于在DNA中靶向和修改腺嘌呤。然后将ecTadA突变体与nCas9 (D10A)融合, 由此标志着第一代腺嘌呤碱基编辑器的诞生, 但最初编辑效率仅为3.2% (Nishida et al., 2016)。为提高腺嘌呤碱基编辑系统的编辑效率, 研究人员构建了一个全面的TadA序列突变文库, 然后将其与dCas9蛋白融合。该方法能够利用抗生素进行抗性筛选, 故而提高了效率。此外, 通过定向进化方法, 包括易错PCR和DNA重组, 进行了7轮迭代进化, 成功开发出ABE7.10。此版本显著提高了基因编辑的效率和精度。值得注意的是, ABE7.10在靶向基因组的特定区域, 特别是在sgRNA的第4-7位置表现出明显的高效性(Yan et al., 2018; Koblan et al., 2018)。随后, 在ABE7.10的基础上, 研究人员转向进一步优化腺嘌呤脱氨酶, 旨在提高碱基编辑效率。通过对TadA7.10的定向优化, 成功研发出TadA8e (Gaudelli et al., 2020; Richter et al., 2020)。从ABE8e到ABE9的进化是通过对TadA8e进行增强实现的。科研人员将2个突变L145T和N108Q精准地引入到TadA-8e中, 并依托冷冻电子显微镜(cryo-EM)进行深入分析, 显著提升了ABE9在编辑窗口的活性, 特别是在sgRNA的5-6位置处, 从而大幅提高了基因编辑精度(Chen et al., 2023)。此外, ABE9还能消除对胞嘧啶的非目标编辑, 降低非靶基因编辑的发生概率。
CBE和ABE碱基编辑系统能够执行4种类型的碱基转换, 即C-T、G-A、A-G和T-C。然而, 对于其它8种可能的碱基转换(C-A、C-G、G-C、G-T、A-C、A-T、T-A和T-G)以及碱基插入和删除能力不足, 这通常对于纠正更广泛的遗传异常至关重要。为解决此限制, David Liu团队将Cas酶、M-MLV-RT逆转录酶以及特别设计的引导RNA (pegRNA)进行整合, 即引导编辑(PE)。此创新方法可实现对基因组的精准编辑, 包括精确的插入、删除以及全面的碱基转换, 覆盖所有12种类型的碱基转换, 且无需依赖DSB或额外的DNA模板, 极大地提升了基因编辑的效率和准确性(图2C) (Doman et al., 2020)。该系统巧妙地利用sgRNA精准地引导逆转录酶到达特定的DNA位点, 之后逆转录酶直接启动并驱动pegRNA中嵌入的模板进行逆转录, 将其精准地转录到DNA链上, 从而实现高度目标导向且精确的遗传修饰(Doman et al., 2020)。Nelson等(2022)在pegRNA的3'端加入结构RNA基序, 生成epegRNA, 可防止3'延伸的降解, 从而提高了PE系统的编辑效率。同时, 开发出pegLIT工具识别并计算pegRNA与3'基序之间的非干扰核苷酸, 可优化3'结构基序的选择。第一代PE系统将M-MLV-RT与一个切割型Cas9 (nCas9 H840A)融合, 建立了Prime Editing的初始平台。在此基础上, PE2通过进化M-MLV逆转录酶扩大PE的潜力, 显著提高了系统生成靶向突变和插入缺失的效率。Bai等(2024a)受DNA靶序列染色质开放性显著影响基因编辑效率的启发, 在基因编辑载体中共表达促进染色质开放的人源RNA m6A去甲基化酶hFTO, 显著提高了编辑效率。随后的PE3又有所创新, 即使用额外的sgRNA设计用于切割非互补DNA链, 在某些特定细胞环境下(如293T细胞)其编辑效率高达78% (Haapaniemi et al., 2018; Grünewald et al., 2019; Jin et al., 2019; Zhou et al., 2019; Zuo et al., 2019; Kurt et al., 2021)。Chen等(2021)对影响引导编辑效果的内源性因子进行了系统筛选, 发现DNA错配修复通路(MMR)起关键作用; 此外, MMR通路的关键因子MLH1的显性负性突变体(MLH1 dn)能增强PE的编辑效率。优化的PE系统融合蛋白结构同样提升了编辑效率, 最终开发出更强大的PE升级版本PE4/PE4max和PE5/PE5max。Qiao等(2023)发现编辑效率低是玉米中应用基因编辑技术的主要障碍。通过设计pegRNA和优化PE蛋白, 显著提高了编辑效率。优化的ePE5max系统具有足够的效率, 能在玉米靶基因中产生可遗传突变, 消除了使用PEs的主要障碍。Liu等(2024)通过暂时抑制MMR系统来优化基因编辑效率, 既提高了编辑效率, 又规避了长期抑制可能引发的基因组不稳定风险。采用优化的PE系统ePE5c, 结合RNA干扰技术可提升编辑效率。Doman等(2023)通过噬菌体辅助进化和蛋白质工程技术, 成功开发出体积更小、效率更高的主要编辑器PE6a和PE6b。在无DNA双链断裂以及额外DNA模板的情况下, 实现靶位点的精准编辑。Yan等(2024)利用全基因组CRISPRi筛选技术, 发现了一个关键的细胞决定因子, 即小RNA结合外切核酸酶保护因子La, 其对于引导编辑至关重要。通过将La与引导编辑蛋白融合, 构建了引导编辑器PE7, 显著提升了编辑效率。
本文的其它图/表
|