
|
|
||
|
基因编辑技术在玉米中的研究进展
植物学报
2024, 59 (6):
978-998.
DOI: 10.11983/CBB24080
基因编辑技术已成为现代农业育种领域的重要工具。玉米(Zea mays)是全球最重要的粮食作物之一, 基因编辑技术在玉米中的应用显示出巨大的潜力。该文综述了基因编辑技术在玉米研究中的应用进展, 重点介绍了CRISPR/Cas等系统在玉米基因组编辑中的最新成果。首先, 介绍了基因编辑技术的基本原理和类型, 特别是CRISPR/Cas系统的工作机制及其在玉米中的应用优势。其次, 总结了基因编辑技术在玉米育种中的研究进展, 涵盖从基础的基因组编辑到复杂的多基因编辑, 旨在改良玉米的产量、品质和抗逆性等关键性状。最后, 文章列举了我国在玉米基因编辑方面的杰出工作, 并讨论了基因编辑技术应用于玉米育种中存在的问题, 同时展望了未来发展方向。 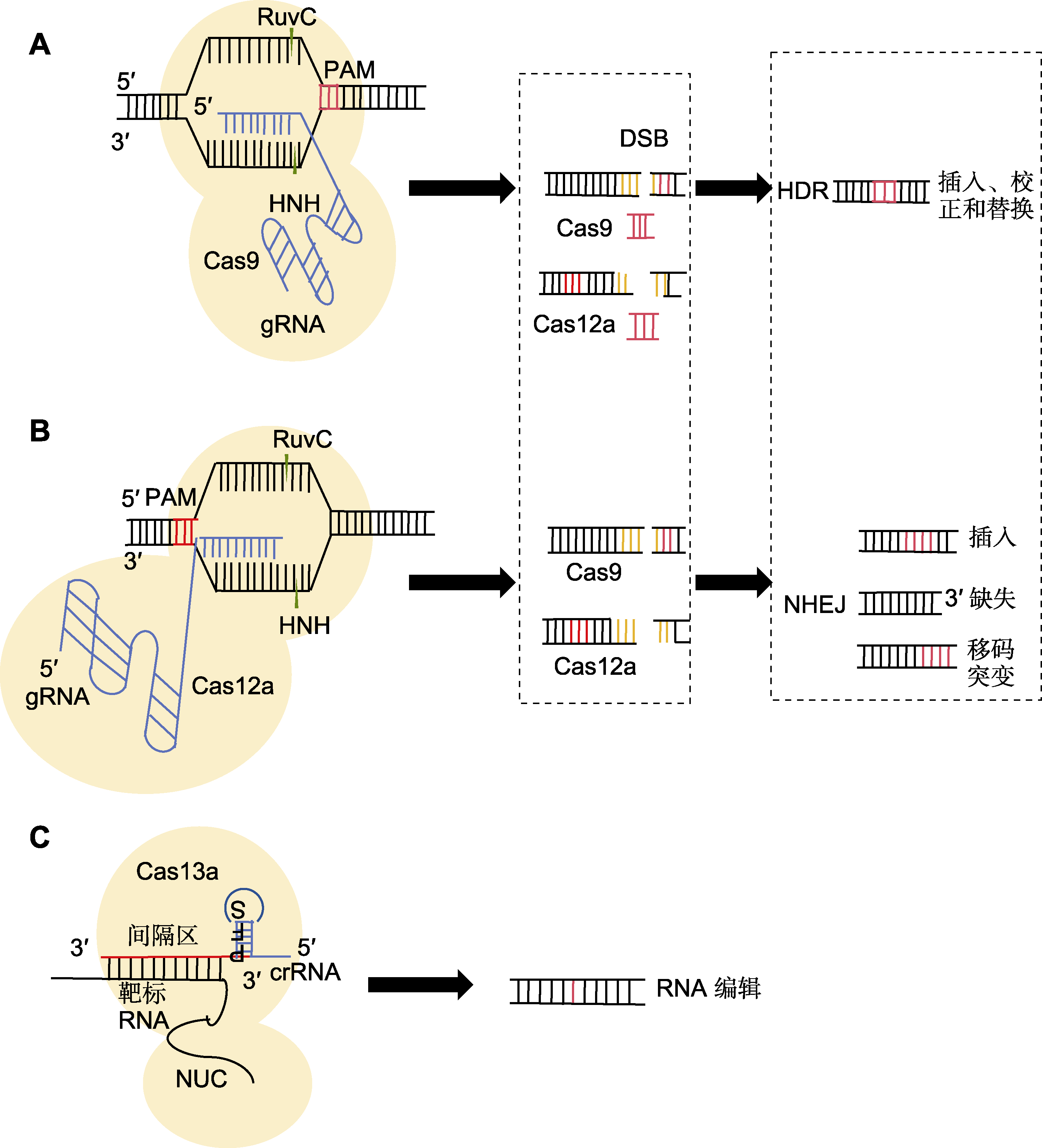 View image in article
图1
CRISPR/Cas基因编辑系统工作原理
(A) Cas9蛋白利用结构域RuvC在原间隔区邻近基序(PAM)序列附近的特定单链DNA上切割, 随后, HNH的结构域与sgRNA配对的DNA另一条单链进行切割形成双链断裂(DSB), 最终进行同源定向修复(HDR)或非同源末端连接(NHEJ)修复成双链; (B) Cas12a通过crRNA介导识别5'-TTTN或5'-TTN的PAM序列; (C) 当crRNA与靶标RNA通过碱基互补配对结合后, 形成crRNA-Cas13复合物, 导致Cas13蛋白构象发生变化, 从而激活其RNA切割活性, 使其能够特异性识别并结合靶标RNA进行切割
正文中引用本图/表的段落
CRISPR/Cas9基因编辑系统由向导RNA (guide RNA, gRNA)和核酸内切酶Cas9复合体构成。Cas9蛋白利用其独特的2种核酸结构域RuvC和HNH精确切割DNA双链。首先, RuvC结构域负责在PAM (protospacer adjacent motif)序列附近的特定单链DNA上执行切割。随后, HNH结构域则对与sgRNA (单链向导RNA)配对的DNA区域的另一条单链进行切割(Jinek et al., 2014)。Jinek等(2012)研究显示, 通过对sgRNA进行精确设计, Cas9能够识别5'-NGG-3'的PAM序列, 并与目标DNA进行互补配对, 引导Cas蛋白对DNA链进行精确切割(图1A), 这为CRISPR/ Cas9系统在植物科学领域的应用奠定了理论基础。2013年, CRISPR/Cas9系统应用于人类细胞编辑, 并成功实现基因编辑和基因突变(Mali et al., 2013; Cong et al., 2013)。此后, CRISPR/Cas系统迅速应用于植物, 并在水稻、小麦(Triticum aestivum)、玉米及拟南芥(Arabidopsis thaliana)等多种植物中实现了特定基因位点的精确突变, 为农业生物技术领域带来了前所未有的突破(Shan et al., 2013; Li et al., 2013; Nekrasov et al., 2013)。
CRISPR/Cas9系统可供选择的靶基因编辑位点相对有限, 同时存在较高的脱靶风险。相比之下, CRISPR/ Cas12系统可提供更广泛的靶向范围, 因此有望成为新一代基因编辑技术。杨帆和李寅(2017)报道的CRISPR DNA核酸内切酶Cpf1, 也称为Cas12a, 其分子较小, 便于在细胞内传递。Cas12a的功能依赖于RuvC和NUC核酸结构域, 并在crRNA的引导下通过识别5'-TTTN或5'-TTN的PAM序列, 对目标DNA进行精确切割, 从而产生DNA双链断裂(Shmakov et al., 2015)。CRISPR/Cas12系统在精确性和安全性方面较CRISPR/Cas9系统具有明显优势。例如, 在CRISPR/Cas9系统中, Cas9蛋白通过识别PAM序列对靶基因进行切割, 但当sgRNA解旋后, Cas蛋白可能错误识别并结合与PAM序列类似的非靶基因, 从而导致脱靶, 且这种错误结合不可逆。相比之下, Cas12蛋白结合在非靶基因时形成一个半封闭的R-环, 只有在识别到真正的PAM序列时才会转变为一个封闭的R-环, 这种可逆的结合方式显著降低了脱靶率。此外, Cas12a蛋白具有双重酶活性, 能同时切割DNA和RNA, 且使用的crRNA长度不到sgRNA的一半, 这使Cas12a系统在细胞中的运送更高效。Cas12a与Cas9的切割机制有所不同。当DNA被切割后, Cas12a形成有5个核苷酸(nt)的黏性末端, 这种特殊的末端结构更倾向于促进HDR过程, 因此显著提高了基因编辑的精确性。Cas9则生成平直的末端, 且其切割位点与PAM序列的距离较远, 这可能导致在NHEJ过程中发生重复识别和切割, 进而增加了基因编辑的不确定性(图1B) (Bandyopadhyay et al., 2020)。
CRISPR/Cas13系统是已鉴定并应用于植物的新型基因编辑技术。该系统由crRNA和核酸酶组成, 并含有2个HEPN (高等真核生物和原核生物核苷酸结合内切酶)结构域, 这使其能够在RNA水平上进行编辑。CRISPR/Cas13系统含有4种不同类型的Cas蛋白, 即Cas13a、Cas13b、Cas13c和Cas13d (Cox et al., 2017)。在CRISPR/Cas13系统的多个亚型中, Cas13a表现出显著优势。其分子较小, 易于构建表达载体和导入植物细胞。Cas13a由1个单独的crRNA分子进行引导, 该crRNA通过与靶RNA进行碱基互补配对实现相互作用, 并且此过程诱导Cas13a发生构象变化。故而Cas13a能够结合crRNA, 并特异性地顺向切割目标单链RNA。更为独特的是, 在完成对目标RNA的顺向切割后, Cas13a还表现出反式切割活性, 即能够不受限制地继续切割其它非靶标RNA分子(Freije et al., 2019)。不同于Cas9和Cas12蛋白, Cas13蛋白可在切割靶RNA后仍然保持活性, 并继续切割其它非靶RNA序列, 从而导致更多非靶序列被破坏(图1C)。CRISPR/Cas13系统已应用于敲除哺乳动物细胞和植物中基因表达和RNA检测。在Aman等(2018)的研究中, CRISPR/Cas13系统成功应用于萝卜(Raphanus sativus)花叶病毒(Turnip Mosaic Virus, TuMV)的干扰。Gootenberg等(2017)将Cas13蛋白改造为一种低成本、高效率及高灵敏度的诊断工具, 即SHERLOCK (specific high-sensitivity enzymatic reporter unlocking), 通过追踪控制靶向RNA的位置和Cas13蛋白的切割, 使CRISPR系统的灵敏度大幅提高, 几乎在所有环境下都可使用。
气孔运动通过调节CO2的可用性影响植物的光合速率, 这一过程受到光合产物的反馈调节。然而, 在单子叶作物中, 该调节回路的遗传组分和机制尚待进一步阐明。Wang等(2019b)基于图位克隆CST1并通过CRISPR/Cas9系统, 发现导致CST1蛋白E81K突变的是一种属于Clade I SWEET (Sugars Will Eventually be Exported Transporters)家族的基因突变。CST1编码一个在附属细胞中表达的功能性葡萄糖转运蛋白。E81K突变破坏了CST1蛋白的寡聚化能力及其葡萄糖转运活性, 导致气孔开度减小、碳饥饿以及叶片提前衰老。此外, 一个含有DYW基序的五肽重复蛋白(pentatricopeptide repeat protein, PPR)参与叶绿体NADH脱氢酶复合体ndhB亚基C到U的编辑。Huang等(2020)利用CRISPR/Cas9技术对qkw9基因进行敲除, 在qkw9的空白背景下, ndhB的C到U编辑被抑制, 光合作用减弱。作物产量的形成依赖于从叶片到种子的蔗糖高效分配。ZmSWEET13旁系同源体(Zmsweet13a、Zmsweet13b和Zmsweet13c)是叶片中表达量最高的基因之一。Bezrutczyk等(2018)通过基因编辑进行三重敲除, 突变体表现严重的生长受阻, 光合机构受损, 叶片积累大量可溶性糖和淀粉, 且RNA-seq数据揭示突变体中与光合作用和碳水化合物代谢相关的基因发生转录失调。ANT (AINTEGUMENTA)是一种AP2转录因子, 在植物生长和花器官形成中起重要作用。Liu等(2020)利用CRISPR/Cas9技术生成1个单密码子缺失突变体和2个ANT1同源基因移码突变体。这2个移码突变体与野生型(WT)相比, 表现出植物光合效率和生长速率降低及叶面积变小。在高密度种植条件下, 玉米植株之间相互遮挡, 导致底部叶片光照强度降低, 进而影响红光与远红光的比例, 最终降低产量。为解释该现象的分子机制, Wu等(2019)利用CRISPR/Cas9技术对玉米中参与光信号传递和光依赖形态建成的PIF基因家族成员Zmpif3、Zmpif4和Zmpif5进行敲除, 发现在黑暗条件下生长的突变体幼苗的中胚轴伸长被严重抑制, 且对模拟遮荫处理的响应减弱。
本文的其它图/表
|