
|
|
||
|
重要林木樟科植物全基因组测序研究进展
植物学报
2024, 59 (2):
302-318.
DOI: 10.11983/CBB23035
近年来, 随着测序技术的革新、测序成本的降低和生物信息学软件的开发, 植物全基因组研究蓬勃发展。樟科(Lauraceae)隶属被子植物木兰类, 泛热带分布, 物种多样性高, 其中很多物种具有重要的经济和生态价值, 目前已发表包括8个物种的13个基因组。该文从樟科全基因组研究现状、基因组特征、起源和进化以及功能基因和基因家族4个方面进行综述, 着重介绍基于组学数据的木兰类及樟科的系统发生、樟科经历的多倍化事件以及与樟科花器官进化和代谢产物相关的基因鉴定。结合研究现状展望了樟科基因组研究的发展方向, 建议通过增加测序基因组分支的代表性并关注具有特殊价值的物种, 及研究物种特异性功能基因以加深对该家族基因功能和进化的理解。 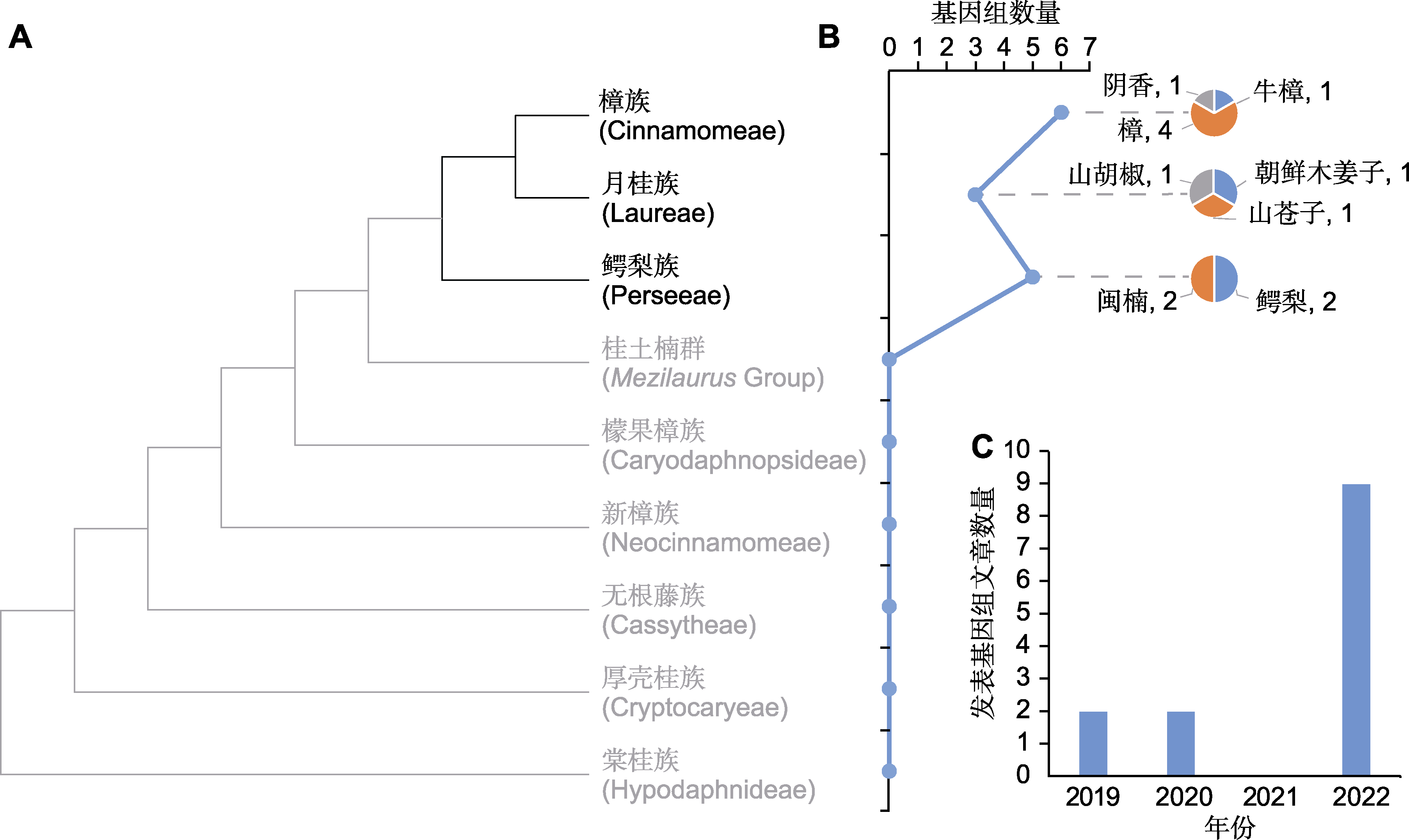 View image in article
图2
樟科基因组已测序物种概况
(A) 樟科系统发生树(参考Liu et al.,
正文中引用本图/表的段落
自2019年樟科第1个全基因组, 即牛樟(Cinnamomum kanehirae=Camphora kanahirae) (Chung and Hsieh, 2023)的基因组序列发表以来(Chaw et al., 2019), 樟科全基因组研究呈现蓬勃发展的态势。目前已有8个物种完成测序(图2; 表1), 分布于樟科3个族(3/9) 6个属(约6/50)。牛樟基因组不仅是樟科首个完成全基因组测序的物种, 也是木兰分支第1个测序的物种(Chaw et al., 2019)。该研究从全基因组层面探讨了木兰类与双子叶植物的关系, 发掘了与牛樟形成相关的基因。随后, 在2019-2020年, 樟科重要的水果鳄梨(Persea americana)、化学物质原料山苍子(Litsea cubeba)以及优良木材树种闽楠(Phoebe bournei)的基因组相继发表(Rendón-Anaya et al., 2019; Chen et al., 2020a, 2020b)。这些物种基因组丰富了樟科基因组数据库, 为樟科基因组研究奠定了基础。
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al., 2022a)。仅一年间共有9篇樟科基因组文章发表(图2C; 表1), 樟(Camphora officinarum) (Jiang et al., 2022; Shen et al., 2022; Sun et al., 2022a; Wang et al., 2022)、阴香(Cinnamomum burmanni) (Li et al., 2022)、朝鲜木姜子(Litsea coreana) (Zhang et al., 2022)及山胡椒(Lindera glauca) (Xiong et al., 2022)的全基因组序列相继发布, 鳄梨和闽楠的基因组质量得到提升(Han et al., 2022; Nath et al., 2022)。尤其值得关注的是, 樟作为我国重要的经济树种, 2022年有4个高质量基因组由不同单位主导发表(图2B; 表1)。
樟科基因组研究中鉴定出3次多倍化事件(图2A)。在樟科所有测序基因组中都检测到木兰目和樟目分化之前发生的1次WGD事件(约118-147 Ma) (Sun et al., 2022a; Han et al., 2022)以及已测序樟科物种共同祖先中发生的1次WGD事件(约76-95 Ma) (Shen et al., 2022; Han et al., 2022)。较近的樟科WGD事件与晚白垩纪早期樟科植物随着冈瓦纳大陆的分裂快速辐射分化的时间一致(Chanderbali et al., 2001)。WGD事件通过改善基本生理活动和初级代谢使植物对环境变化的适应性增强, 促进了早期樟科物种的快速辐射(Chen et al., 2020a; Jiang et al., 2022; Sun et al., 2022a)。值得注意的是, 山胡椒基因组在与相近的山苍子分化后(14.90-23.18 Ma)独自经历了1次多倍化事件, 与早中新世(23 Ma)月桂族快速辐射分化的时间一致(Qin et al., 2023)。月桂族为樟科中雌雄异株类群, 主要分布在亚洲热带和亚热带地区, 为亚热带常绿阔叶林的建群种(Li et al., 2004; Qin et al., 2023)。这次多倍化事件导致山胡椒具有比山苍子及其它樟科植物大将近1倍的基因组和更多的蛋白质编码基因(表1) (Xiong et al., 2022)。其中, 与萜烯合酶生物合成相关的基因家族发生显著扩张, 可能对山胡椒的生态适应性和生物适应性有重要作用(Pichersky and Raguso, 2018; Xiong et al., 2022), 有利于其应对早中新世东亚季风盛行引起的极端天气(Qin et al., 2023)。
总体来说, 樟科基因组研究呈现3个明显的特点。(1) 樟科基因组特征信息十分匮乏。除樟族、月桂族和鳄梨族以外的樟科物种基因组大小未见报道(图2; 表1)。(2) 已测序物种明显地向中国分布及有经济价值的物种倾斜。首先, 测序物种主要集中在樟和闽楠等作为化学物质原料或提供优质木材的经济树种。其次, 2019年以来一直是我国学者在主导樟科基因组研究, 已发表的基因组中, 仅鳄梨基因组由国外学者报道, 其余全部为中国学者对国产种类的研究报道。此外, 已发表的樟科基因组集中在核心樟类群(core Lauraceae), 对樟科原始分支棠桂族、厚壳桂族、新樟族、檬果樟族及无根藤族等缺少研究(图2)。最后, 在空间分布上, 测序种类表现出明显的亚洲分布倾向, 作为樟科分布中心的热带非洲和南美洲物种(除鳄梨外)未有涉及, 这也与参与樟科基因组研究的中国学者居多有关。(3) 功能基因的挖掘不够深入。目前, 学者们侧重于挖掘重要的功能基因并解析其分子机制, 但樟科基因组研究还停留在基因层面, 缺少功能基因的验证及具体机制探究, 除TPS基因家族在樟科的起源与进化得到探讨外(Han et al., 2022), 对功能基因的系统研究和比较分析十分缺乏(图6)。此外, 与花器官进化、木材形成和代谢产物合成相关的功能基因是研究热点, 而樟科作为被子植物基部类群, 其经历漫长的历史变迁仍在森林中占据优势的独特生物学特性尚未得到足够的关注(图6)。
(2) 增加测序基因组分支的代表性。目前樟科测序的基因组仅涉及3个族, 还有6个族尚无代表物种的基因组发表(图2; 表1)。樟科具有复杂的进化历史, 从系统发生拓扑结构不一致中可窥探一二。有代表性的物种取样能够帮助厘清樟科的系统发生关系。注释良好的基因组数据为使用重测序和靶向捕获的方法从标本中获取大量的基因序列提供参考, 这将解决樟科采集和鉴定难题, 从而构建更具有代表性的系统发生树(Sun et al., 2022a)。一方面, 解析樟科系统发生关系不仅可帮助我们从进化角度探讨特定功能基因的生物学特性和分子机制, 还能为保护濒危植物提供理论依据。桫椤(Alsophila spinulosa)基因组就是1个良好的案例(Huang et al., 2022)。研究者基于9个群体107个桫椤个体的基因组重测序解析树蕨类植物树干形成发育的独特性, 为理解蕨类植物的进化提供了新视角, 也从影响桫椤种群变化的因素中拓展了保护工作思路。另一方面, 基于系统发生树和分布数据可揭示樟科的时空分布格局, 解析其类群多样分布类型的成因。例如, 泛热带分布的厚壳桂属和琼楠属(Beilschmiedia) (Chanderbali et al., 2001), 东亚-北美间断分布的檫木属(Sassafras) (Yang et al., 2022b; Qin et al., 2023), 亚洲-大洋洲分布的土楠属(Endiandra) (Li et al., 2020), 以及仅分布在亚洲的新樟属(Neocinnamomun)和仅分布在美洲的桂土楠群(Chanderbali et al., 2001; Liu et al., 2021)。此外, 大量遗传信息和代表性取样能够为解析樟科复杂的进化历史提供支持(Liu et al., 2021; Tian et al., 2021)。目前, 樟科科内的多倍化只在山胡椒基因组中检测到(图5), 考虑到月桂族多样的染色体变化及其更大的基因组(图3), 增加月桂族物种全基因组测序, 有助于理解多倍化在樟科进化中的作用。
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
2022年是樟科基因组研究成果爆发之年, 三代测序技术的发展特别是PacBio公司循环共识测序模式(circular consensus sequencing, CCS)生成HiFi reads (high fidelity reads)的技术促进了樟科全基因组测序研究(Sun et al.,
本文的其它图/表
|