番茄斑萎病毒(tomato spotted wilt virus, TSWV)隶属布尼亚病毒目(Bunyavirales)番茄斑萎病毒科(Tospoviridae)正番茄斑萎病毒属(Orthotospovirus) (Peiró et al., 2014)。TSWV分子基因组由3条RNA单链构成, 分别为L RNA、M RNA和S RNA (Adkins, 2000)。L RNA是反义RNA链, 长约8.9 kb, 包含1个开放阅读框(ORF), 编码331 kDa的RNA依赖性聚合分子蛋白(De Haan et al., 1991); M RNA含2个ORF, 长约4.8 kb, 是双义RNA链, 分别编码34和127.4 kDa的运输蛋白和糖蛋白(Kormelink et al., 1992); S RNA含2个ORF, 长约2.9 kb, 是双义RNA链, 分别编码54和29 kDa的沉默抑制子和核衣壳蛋白N (Nucleocapsid) (De Haan et al., 1990)。
番茄斑萎病毒因第1次在番茄(Lycopersicon esculentum)上致病被人类发现得以正式命名。TSWV 寄主范围广, 目前已有100多个科1 090多种植物遭受该病毒侵染(吴鑫本, 2008)。TSWV在蓟马幼虫体内完成复制增殖后, 以蓟马成虫为媒介进行病毒传播。西花蓟马(Frankliniella occidentalis)是所有蓟马中病毒传播效率最高的种类(邱树亮等, 2012)。在不同生育期番茄受TSWV侵染后表现出的发病症状不同。苗期会引起生长点和幼嫩叶片颜色变为铜色并使叶片呈上卷状, 随后出现黑色环状病点和黑褐色斑块, 叶背面沿叶脉部位逐渐变紫, 茎端出现一些看似黑褐色的坏死条斑, 番茄植株矮化, 生长速度变缓, 并出现萎蔫等不良病症; 坐果期果实表面会出现淡绿色环形斑块, 表面伴有轻微凸起, 并有一些细微轮纹出现; 如在青果期发病, 果实表面则会出现褐色坏死斑块, 并似瘤状突起, 导致果实极易脱落; 果实成熟期轮纹更加明显, 出现许多红白相间或红黄相间的轮纹斑块, 严重时整个果实呈褐色坏死状, 且果实脐部的症状类似脐腐病。果实完全成熟时, 上述环状病点和黑褐斑块的环状轮纹会更加明显, 发病的果实表皮最终会变成黑褐色, 并坏死(Chiemsombat and Adkins, 2006)。TSWV最早在20世纪初被澳大利亚研究者发现(Brittlebank, 1919), 后来在美国夏威夷、西班牙、阿根廷、南非和巴西等地流行并对当地番茄生产造成严重的经济损失(Soler et al., 2003)。2003年, 在我国北京首次检测出TSWV (张友军等, 2003), 随后在云南等地的多种园艺作物上发现了TSWV, 同时发现蓟马的危害范围也在进一步扩大(丁铭等, 2004; 饶雪琴等, 2010)。2012年, 在山东寿光、云南昆明和北京顺义等地相继出现TSWV危害(邱树亮等, 2012), 一些大棚和温室的番茄生产受损严重, 损失量约为80%, 有的甚至出现绝收。
由于TSWV的危害日趋严重, 2011年该病毒被列入危害性强的十大重要植物病毒清单(Scholthof et al., 2011)。至今, 银川地区还没有该病毒鉴定的相关报道。我们在对银川番茄病毒病进行调查时, 发现一些番茄植株可能遭受了TSWV侵染, 在大部分温室中发病率约5%, 露地发病率约3%; 严重时发病率高达60%-70%。为明确银川市番茄是否已遭受TSWV危害, 本研究利用TSWV的国家标准检测引物, 通过RT-PCR和Western blot对银川市宁夏大学实验农场的14份番茄病毒病疑似样本进行检测, 以期为宁夏设施番茄抗番茄斑萎病种质鉴定和品种选育提供理论依据。
1 材料与方法
1.1 材料
2020年7-9月, 从宁夏回族自治区银川市永宁县宁夏大学实验农场6号日光温室内, 采集番茄(Lycopersicon esculentum Mill.)材料62386-7-0疑似感染TSWV病毒病叶片样本14份, 于-80°C冰箱保存。TSWV抗体由南京农业大学陶小荣教授惠赠(兔多抗, 效价1:10 000)。
1.2 病叶样本总RNA的提取及反转录
从-80°C超低温冰箱中取出保存的14份病叶样本, 放入研钵中并加入液氮, 快速研磨至呈粉末状, 使用Trizol法提取样本总RNA (程国新, 2019), 然后检测总RNA质量, 最后利用试剂盒进行反转录。反应结束后, 将产物放入-20°C冰箱, 备用。
1.3 TSWV检测
根据已报道的番茄斑萎病毒病RT-PCR检测方法(中华人民共和国国家质量监督检验检疫总局和中国国家标准化管理委员会, 2013)国家标准, 合成引物TSWVN-F: 3′-GCTTTGTTGACACAAGGCAAAGAC C-5′; TSWVN-R: 3′-GGCAAGCCTCACAGACTTTG CATC-5′ (退火温度58°C, 片段长度约390 bp)。检测14份病叶样本的cDNA浓度。PCR反应体系(25 μL)及反应程序详见番茄斑萎病毒病RT-PCR检测方法(中华人民共和国国家质量监督检验检疫总局和中国国家标准化管理委员会, 2013)。用通用型TSWV基因试剂盒检测的确定受TSWV侵染的番茄叶片作为阳性对照, 以生长势强且健康的番茄叶片为阴性对照。将扩增得到的PCR产物进行电泳检测。
1.4 TSWV基因克隆、测序与多序列比对分析
回收纯化目的片段, 连接载体后转化大肠杆菌(Escherichia coli)感受态, 然后将菌液均匀涂布于含有氨苄的LB培养基上, 37°C倒置过夜培养。随机挑取平板上的单克隆进行菌落RT-PCR检测, 筛选阳性克隆进行测序。得到测序结果之后, 利用NCBI网站对基因序列进行载体序列去除, 然后利用BLAST (
1.5 TSWV的Western blot检测
对RT-PCR检测结果为阳性的样本进一步进行Western blot检测(Tamura et al., 2011)。取约0.1 g新鲜组织样本, 液氮速冻研磨呈粉末状, 将蛋白质提取液加入提前预冷的2 mL离心管中与粉末样本混匀, 沸水浴5分钟, 离心机15 294 ×g离心5分钟后取上清(即蛋白质)进行聚丙烯酰胺凝胶电泳; 在甲醇中激活PVDF膜; 随后取出PVDF膜, 用去离子水洗涤后浸入封闭液中, 于摇床上封闭90分钟, 缓慢摇动; 取出膜, 浸入TBST稀释的TSWV抗体中孵育(25°C, 1.5小时); 洗涤后转入含二抗的玻璃皿中, 在摇床上孵育1-1.5小时; 取出后再洗涤3次。然后, 将其浸入缓冲液中平衡5分钟, 置于保鲜膜上, 取适量ECL发光试剂混匀后加在膜表面, 移入凝胶成像分析仪, 观察结果。
2 结果与讨论
2.1 田间病株症状
图1
图1
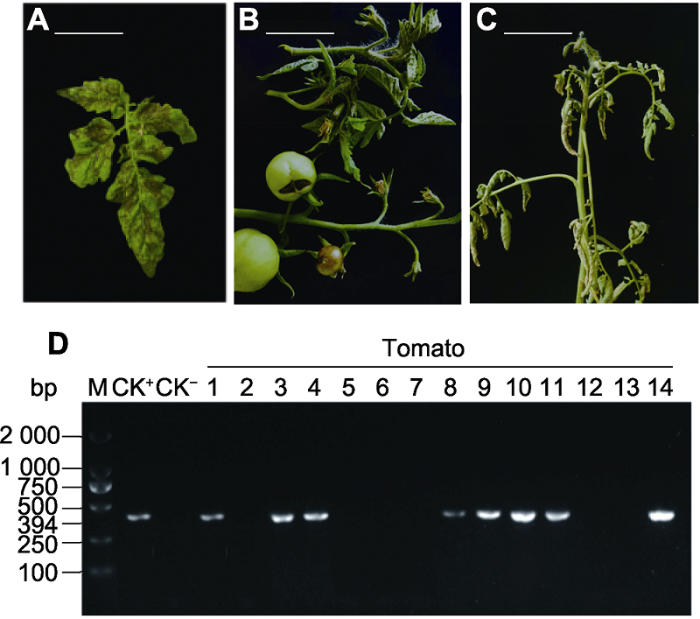
番茄田间采集病样症状及TSWV的RT-PCR检测
(A) 叶片出现黑斑; (B) 果实伴有黄化和畸形; (C) 植株萎蔫; (D) 病样TSWV的RT-PCR检测(M: DL2000 marker; CK+: 阳性对照; CK-: 阴性对照; 1-14: 番茄病叶样本)。Bars=10 cm
Figure 1
RT-PCR detection of TSWV and disease symptoms in tomato field
(A) Black spots appeared on the leaves; (B) The fruits sho-wing yellowing and deformity; (C) Wilted plant; (D) Detection of TSWV in disease samples by RT-PCR (M: DL2000 mar-ker; CK+: Positive control; CK-: Negative control; 1-14: To-mato diseased leaf samples). Bars=10 cm
2.2 RT-PCR鉴定结果
对采集的样本进行总RNA提取, 利用引物TSWVN-F和TSWVN-R进行RT-PCR检测, 14份番茄病样中有8份样本检测出与预期N基因序列目的条带大小(约390 bp)相近的片段, 阴性对照则未扩增出任何条带(图1D), 表明这8份样本中伴有TSWV感染。
2.3 TSWV基因克隆、多序列比对及系统进化分析
利用试剂盒回收扩增到的8个目的片段, 通过菌落PCR筛选阳性克隆, 并送公司测序, 结果显示8条序列完全一致。
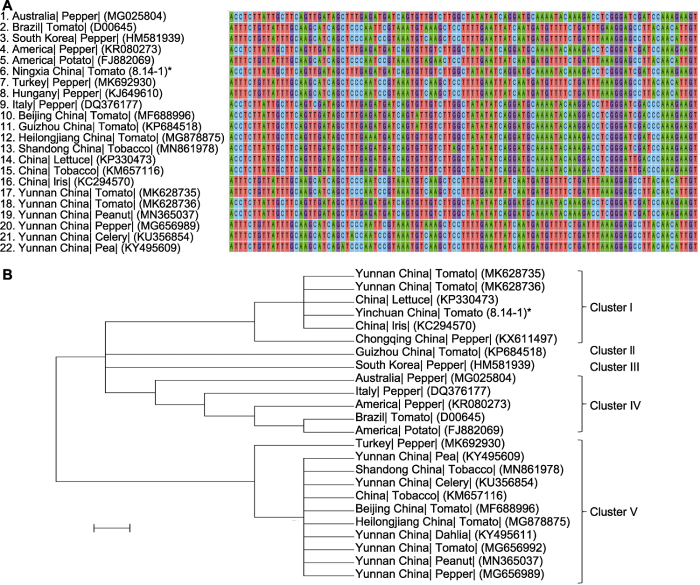
本研究克隆到的N基因序列全长为394 bp, 编码131个氨基酸残基。BLAST分析结果显示, 其与番茄TSWV的N基因核苷酸序列相似性达99.49%。利用MEGA 5.0软件将本研究所得的TSWV N基因序列与已报到的6个国外和10个国内TSWV的N基因序列进行多序列比对(图2A), 发现其与美国辣椒(Capsicum annuum) (KR080273)、中国黑龙江番茄(MG-878875)、中国云南番茄(UK528736)和中国云南花生(Arachis hypogaea) (MN365037)中的TSWV分离物N基因序列完全一致。
图2
图2
基于N基因序列一致性的片段多序列比对(A)和系统进化树分析(B)
*: 本研究N基因序列。Bar=0.005
Figure 2
Multiple sequence alignment (A) and phylogenetic tree analysis (B) based on sequence consistency of N gene
*: N gene sequence of this study. Bar=0.005
系统进化分析(图2B)发现, TSWV在N基因序列的系统进化树上分为5个簇, 其中中国分离物主要聚集在第1个簇(Cluster I)和第5个簇(Cluster V), 澳大利亚、美国、意大利和巴西等分离物构成第4个簇(Cluster IV), 中国贵州番茄分离物和韩国辣椒分离物则分别构成第2个簇(Cluster II)和第3个簇(Cluster III)。
本研究采集的银川分离物聚集在第1个簇, 与云南番茄(MK628735和MK628736)、中国莴苣(Lactuca sativa) (KP330473)、中国鸢尾(Iris tectorum) (KC294570)和重庆辣椒(KX611497)的TSWV分离物相对近缘, 与山东、黑龙江和北京等地及国外的分离物相对远缘, 上述结果表明, 本研究从银川番茄病样上检测到的病毒为TSWV。
2.4 TSWV的Western blot检测
基于RT-PCR检测结果, 将8份阳性样本1、3、4、8、9、10、11和14进行总蛋白提取, 进一步通过Western blot检测验证。结果显示, 8份病样中检测到的条带与预期TSWV N基因蛋白大小(29 kDa)相近(图3)。综上, 本研究的血清学检测结果也表明田间采集的番茄病样中存在TSWV侵染。
图3
图3
番茄斑萎病毒的Western blot检测
M: 分子量蛋白marker; CK-: 阴性对照; 1、3、4、8、9、10、11和14: 检测样本
Figure 3
Western blot analysis of tomato spotted wilt virus
M: Protein size marker; CK-: Negative control; 1, 3, 4, 8, 9, 10, 11 and 14: Test samples
2.5 讨论
TSWV是全世界范围内频繁发生且对植物危害非常严重的一种病毒, 2011年被国际组织列为世界上对植物危害最大的十大病毒之一, 仅次于烟草花叶病毒(tobacco mosaic virus, TMV), 位居第二(Scholthof et al., 2011)。目前, 在我国设施及露地种植作物中TSWV危害日趋严重, 已蔓延至多个省、市, 如黑龙江(番茄) (王凯娜等, 2019)、重庆(辣椒) (孙淼等, 2017)、青海(辣椒) (吴淑华等, 2020)、山东(烟草(Nicotiana tabacum)) (张万红等, 2020)以及北京(莴苣和菊花(Dendranthema morifolium)) (陈东亮等, 2018; 金凤媚等, 2020), 对多种作物造成了严重的经济损失。但银川尚未见关于TSWV的报道。
本研究在对番茄病害开展调查过程中, 在温室和露地栽培的番茄上均发现疑似TSWV的症状。为明确番茄是否受TSWV侵染, 对采自宁夏大学实验农场的14份样本进行了分子鉴定, 并对分子鉴定结果呈阳性的8份样本进行Western blot检测。分子鉴定和蛋白检测结果一致, 证实银川番茄遭受了TSWV侵染。本研究中的TSWV N基因序列与国内16个和国外7个TSWV基因序列的同源性较高。系统进化分析结果显示, 银川TSWV分离物与云南番茄、中国莴苣、中国鸢尾和重庆辣椒的TSWV分离物相对近缘, 与山东、黑龙江和北京等地及国外的TSWV分离物相对远缘。早在20世纪70年代, 研究者发明了酶联免疫吸附法(enzyme linked immunosorbent assay, ELISA), 检测植物是否受到病毒侵染。后来, 因该方法具备所需材料少、操作便捷且能同时测定多种病毒的特点被广泛应用。孙淼等(2017)利用Dot-ELISA及电镜观察法检测出阳性样品, 然后利用分子鉴定进行验证, 在辣椒上检测出TSWV。吴淑华等(2020)在分子鉴定的基础上, 利用TSWV的抗体先进行分子鉴定, 然后通过Western blot对样品进一步检测, 结果证明青海辣椒上存在TSWV感染。受实验室电镜设备等系列条件限制, 本研究首先通过分子鉴定(RT-PCR)检测出阳性样品, 然后通过Western blot鉴定TSWV, 两种鉴定结果完全吻合。PCR技术对实验环境和操作要求较低, 而TSWV属于RNA病毒, RNA提取过程对实验条件要求相对较高; Western blot不仅对实验条件要求较高, 而且出现假阳性结果概率较低。因此, 相比于孙淼等(2017)的检测方法, 本研究与吴淑华等(2020)的研究类似, 将分子鉴定放在第1步, 极大地降低了检测的工作量, 提升了检测效率, 且PCR鉴定技术与Western blot检测相结合得到的结果更加可靠(郭亚璐等, 2017)。
2018年上半年宁夏越夏茬番茄的生产总面积约为5.2×103 hm2, 其中平罗县的栽培面积最大, 为8.67×102 hm2, 上市量约达5.0×105 t, 70%左右的番茄远销上海、广州、四川和重庆等地(李艳莉等, 2019)。因此, 番茄对于当地农民来说不仅是食用蔬菜, 而且是提升收入水平的主要经济作物。TSWV的发生可能会对银川番茄及其它经济作物产量造成难以预估的损失, 在生产上我们要密切关注此类病害的发生, 及时防治。本研究发现, 银川已有TSWV发生, 并预示银川的TSWV可能由其它地区传入, 可对当地经济作物造成危害。此外, 在调查时还发现田间存在大量蓟马群体, 因TSWV是一种虫传植物病毒, 蓟马是重要的传播媒介, 可长期传播。为避免损失, 应树立防患意识, 选育抗病品种, 加强种苗检疫, 严格把控流通环节, 减少带有TSWV病毒种苗进入区内, 同时还要重视田间蓟马的防治, 降低传毒基数, 从而有效控制病毒大范围扩散。
参考文献

Taxonomy: Tomato spotted wilt virus (TSWV) is the type member of the plant-infecting Tospovirus genus in the family Bunyaviridae, a large group of predominantly vertebrate- and insect-infecting RNA viruses. Physical properties: Virions are 80-120-nm pleomorphic particles with surface projections composed of two viral glycoproteins, G1 and G2 (Fig. 1). Virion composition is 5% nucleic acid, 70% protein, 5% carbohydrate and 20% lipid. The genome consists of three negative or ambisense ssRNAs designated S (2.9 kb), M (4.8 kb) and L (8.9 kb), with partially complementary terminal sequences that allow the RNA to adopt a pseudocircular or panhandle conformation. Each genomic RNA is encapsidated by multiple copies of the viral nucleocapsid (N) protein to form ribonucleoprotein structures also known as nucleocapsids. The nucleocapsids are enclosed in a host-derived membrane bilayer along with an estimated 10-20 copies of the L protein, the putative RNA-dependent RNA polymerase. Hosts: Over 800 plant species, both dicots and monocots, in more than 80 plant families are susceptible to TSWV (Goldbach and Peters, 1994). The Solanaceae and Compositae families contain the largest numbers of susceptible plant species (Prins and Kormelink, 1998). TSWV also replicates in its insect vector, thrips (Thysanoptera: Thripidae) (Ullman et al., 1993; Wijkamp et al., 1993). Useful web site: http://www4.ncbi.nlm.nih.gov/ICTVdb/ICTVdB/11050003.htm.

Lasergene's eight modules provide tools that enable users to accomplish each step of sequence analysis, from trimming and assembly of sequence data, to gene discovery, annotation, gene product analysis, sequence similarity searches, sequence alignment, phylogenetic analysis, oligonucleotide primer design, cloning strategies, and publication of the results. The Lasergene software suite provides the functions and customization tools needed so that users can perform analyses the software writers never imagined.

Western blotting is an important technique used in cell and molecular biology. By using a western blot, researchers are able to identify specific proteins from a complex mixture of proteins extracted from cells. The technique uses three elements to accomplish this task: (1) separation by size, (2) transfer to a solid support, and (3) marking target protein using a proper primary and secondary antibody to visualize. This paper will attempt to explain the technique and theory behind western blot, and offer some ways to troubleshoot.

The avirulence determinant triggering the resistance conferred by the tomato gene Sw-5 against Tomato spotted wilt virus (TSWV) is still unresolved. Sequence comparison showed two substitutions (C118Y and T120N) in the movement protein NSm present only in TSWV resistance-breaking (RB) isolates. In this work, transient expression of NSm of three TSWV isolates [RB1 (T120N), RB2 (C118Y) and non-resistance-breaking (NRB)] in Nicotiana benthamiana expressing Sw-5 showed a hypersensitive response (HR) only with NRB. Exchange of the movement protein of Alfalfa mosaic virus (AMV) with NSm supported cell-to-cell and systemic transport of the chimeric AMV RNAs into N. tabacum with or without Sw-5, except for the constructs with NBR when Sw-5 was expressed, although RB2 showed reduced cell-to-cell transport. Mutational analysis revealed that N120 was sufficient to avoid the HR, but the substitution V130I was required for systemic transport. Finally, co-inoculation of RB and NRB AMV chimeric constructs showed different prevalence of RB or NBR depending on the presence or absence of Sw-5. These results indicate that NSm is the avirulence determinant for Sw-5 resistance, and mutations C118Y and T120N are responsible for resistance breakdown and have a fitness penalty in the context of the heterologous AMV system. © 2014 BSPP AND JOHN WILEY & SONS LTD.

 首页
首页