

植物学报 ›› 2019, Vol. 54 ›› Issue (1): 37-45.DOI: 10.11983/CBB18019
陶乃奇1,张斌2,3,刘信凯4,周和达2,钟乃盛4,严丹峰4,张敏1,高继银4,张文驹1,*( )
)
收稿日期:2018-01-18
接受日期:2018-04-28
出版日期:2019-01-30
发布日期:2019-07-31
通讯作者:
张文驹
基金资助:
Naiqi Tao1,Bin Zhang2,3,Xinkai Liu4,Heda Zhou2,Naisheng Zhong4,Danfeng Yan4,Min Zhang1,Jiyin Gao4,Wenju Zhang1,*( )
)
Received:2018-01-18
Accepted:2018-04-28
Online:2019-01-30
Published:2019-07-31
Contact:
Wenju Zhang
摘要: 利用杜鹃红山茶(Camellia azalea)转录组数据和前人研究中获得的12个高多态性的SSR位点, 采用荧光毛细管电泳分析了21个茶花杂交新品种的基因型。结果显示, 12对SSR引物都能获得稳定清晰的扩增产物, 且上述12个SSR位点的基因型能够完全区分上述21个茶花新品种。其中, 来自同一杂交组合后代的多个品种, 基因型间有差异的位点数为2-10个, 来自不同杂交组合后代的品种间, 其基因型有差异的位点数为5-12个。研究结果表明, 上述21个茶花新品种均获得了独特的基因型标记, 能准确地进行品种身份鉴定, 这对以扦插或嫁接扩大生产的茶花品种的鉴定和品种权保护具有重要意义。
陶乃奇,张斌,刘信凯,周和达,钟乃盛,严丹峰,张敏,高继银,张文驹. 利用荧光标记SSR鉴别21个茶花新品种. 植物学报, 2019, 54(1): 37-45.
Naiqi Tao,Bin Zhang,Xinkai Liu,Heda Zhou,Naisheng Zhong,Danfeng Yan,Min Zhang,Jiyin Gao,Wenju Zhang. Identification of 21 New Camellia Hybrid Varieties by Fluorescence-labelled Simple Sequence Repeat Markers. Chinese Bulletin of Botany, 2019, 54(1): 37-45.
| Cross combination No. | New variety No. | Parental types of cross combinations |
|---|---|---|
| ZH-01 | SJ-01 | Var. B (♀)×Var. A (♂) |
| SJ-02 | Var. B (♀)×Var. A (♂) | |
| SJ-03 | Var. B (♀)×Var. A (♂) | |
| SJ-04 | Var. B (♀)×Var. A (♂) | |
| ZH-02 | SJ-05 | Var. C (♀)×Var. A (♂) |
| SJ-06 | Var. C (♀)×Var. A (♂) | |
| SJ-07 | Var. C (♀)×Var. A (♂) | |
| SJ-08 | Var. C (♀)×Var. A (♂) | |
| ZH-03 | SJ-09 | Var. D (♀)×Var. A (♂) |
| SJ-10 | Var. D (♀)×Var. A (♂) | |
| SJ-11 | Var. D (♀)×Var. A (♂) | |
| ZH-04 | SJ-12 | Var. E (♀)×Var. A (♂) |
| ZH-05 | SJ-13 | Var. F (♀)×Var. A (♂) |
| ZH-06 | SJ-14 | Var. A (♀)×Var. G (♂) |
| SJ-15 | Var. A (♀)×Var. G (♂) | |
| SJ-16 | Var. A (♀)×Var. G (♂) | |
| ZH-07 | SJ-17 | Var. A (♀)×Var. H (♂) |
| SJ-18 | Var. A (♀)×Var. H (♂) | |
| ZH-08 | SJ-19 | Var. A (♀)×Var. I (♂) |
| ZH-09 | SJ-20 | Var. A (♀)×Var. J (♂) |
| ZH-10 | SJ-21 | Var. A (♀)×Var. K (♂) |
表1 21个山茶杂交新品种信息
Table 1 Information of 21 new Camellia hybrid varieties in this study
| Cross combination No. | New variety No. | Parental types of cross combinations |
|---|---|---|
| ZH-01 | SJ-01 | Var. B (♀)×Var. A (♂) |
| SJ-02 | Var. B (♀)×Var. A (♂) | |
| SJ-03 | Var. B (♀)×Var. A (♂) | |
| SJ-04 | Var. B (♀)×Var. A (♂) | |
| ZH-02 | SJ-05 | Var. C (♀)×Var. A (♂) |
| SJ-06 | Var. C (♀)×Var. A (♂) | |
| SJ-07 | Var. C (♀)×Var. A (♂) | |
| SJ-08 | Var. C (♀)×Var. A (♂) | |
| ZH-03 | SJ-09 | Var. D (♀)×Var. A (♂) |
| SJ-10 | Var. D (♀)×Var. A (♂) | |
| SJ-11 | Var. D (♀)×Var. A (♂) | |
| ZH-04 | SJ-12 | Var. E (♀)×Var. A (♂) |
| ZH-05 | SJ-13 | Var. F (♀)×Var. A (♂) |
| ZH-06 | SJ-14 | Var. A (♀)×Var. G (♂) |
| SJ-15 | Var. A (♀)×Var. G (♂) | |
| SJ-16 | Var. A (♀)×Var. G (♂) | |
| ZH-07 | SJ-17 | Var. A (♀)×Var. H (♂) |
| SJ-18 | Var. A (♀)×Var. H (♂) | |
| ZH-08 | SJ-19 | Var. A (♀)×Var. I (♂) |
| ZH-09 | SJ-20 | Var. A (♀)×Var. J (♂) |
| ZH-10 | SJ-21 | Var. A (♀)×Var. K (♂) |
| No. | Primer sequences (5′-3′) | Repeat motifs | Ta (°C) | References |
|---|---|---|---|---|
| 478 | F: CAACACCACCAACAAGA | (AAAGG)4 | 53 | Liu et al., 2008 |
| R: GATATGAGATCCGTCCC | ||||
| SSR2 | F: TATTGCCTACGACCATTTCCA | (GA)14 | 56 | Kaundun and Matsumoto, 2002 |
| R: TTTGAGTTCGTTGCCTTCTCT | ||||
| CamsinM11 | F: GCATCATTCCACCACTCACC | (CA)12 | 60 | Freeman et al., 2004 |
| R: GTCATCAAACCAGTGGCTCA | ||||
| CamSSR01 | F: CCAACAAGAATCAGGAAGAG | (AAT)6 | 54 | In this study |
| R: ATCCAACGGTGGTAGACGAG | ||||
| CamSSR02 | F: AGTTCCGCCTCCAGTTTGAC | (ACG)7 | 54 | In this study |
| R: GGACCGAGAGGTAACAGTGG | ||||
| CamSSR03 | F: GCCACTACCCTCTTTACACC | (CAC)7 | 55 | In this study |
| R: TTCTCTTCCTCTTTCTTCCC | ||||
| CamSSR04 | F: ATGTGTTGAGTAGCGAGCGT | (AT)10 | 56 | In this study |
| R: TTGTCCATCTTTATGTAGGG | ||||
| CamSSR05 | F: GCAAACACCAACTGATTACC | (TA)10 | 56 | In this study |
| R: TTCCATACAACTCAACCAAA | ||||
| CamSSR06 | F: GGTTTGGAAAAAGGACACGC | (GCC)7 | 58 | In this study |
| R: AATCTGCCTCTGGTAGTCCG | ||||
| CamSSR07 | F: TCTCATCCCCATCTTTATCC | (TCC)7 | 58 | In this study |
| R: GTTCCCTGCTGCTGTTGTTA | ||||
| CamSSR08 | F: TCACCAGTCACTTTCCCTCC | (AC)10 | 58 | In this study |
| R: CCACCAAAAGGCACAATACC | ||||
| CamSSR09 | F: CATCATCCATCAAACCGTCC | (AT)10 | 58 | In this study |
| R: GAAGGCACATTGGTTCTGGG |
表2 12对SSR引物信息
Table 2 Information of 12 primer pairs in this study
| No. | Primer sequences (5′-3′) | Repeat motifs | Ta (°C) | References |
|---|---|---|---|---|
| 478 | F: CAACACCACCAACAAGA | (AAAGG)4 | 53 | Liu et al., 2008 |
| R: GATATGAGATCCGTCCC | ||||
| SSR2 | F: TATTGCCTACGACCATTTCCA | (GA)14 | 56 | Kaundun and Matsumoto, 2002 |
| R: TTTGAGTTCGTTGCCTTCTCT | ||||
| CamsinM11 | F: GCATCATTCCACCACTCACC | (CA)12 | 60 | Freeman et al., 2004 |
| R: GTCATCAAACCAGTGGCTCA | ||||
| CamSSR01 | F: CCAACAAGAATCAGGAAGAG | (AAT)6 | 54 | In this study |
| R: ATCCAACGGTGGTAGACGAG | ||||
| CamSSR02 | F: AGTTCCGCCTCCAGTTTGAC | (ACG)7 | 54 | In this study |
| R: GGACCGAGAGGTAACAGTGG | ||||
| CamSSR03 | F: GCCACTACCCTCTTTACACC | (CAC)7 | 55 | In this study |
| R: TTCTCTTCCTCTTTCTTCCC | ||||
| CamSSR04 | F: ATGTGTTGAGTAGCGAGCGT | (AT)10 | 56 | In this study |
| R: TTGTCCATCTTTATGTAGGG | ||||
| CamSSR05 | F: GCAAACACCAACTGATTACC | (TA)10 | 56 | In this study |
| R: TTCCATACAACTCAACCAAA | ||||
| CamSSR06 | F: GGTTTGGAAAAAGGACACGC | (GCC)7 | 58 | In this study |
| R: AATCTGCCTCTGGTAGTCCG | ||||
| CamSSR07 | F: TCTCATCCCCATCTTTATCC | (TCC)7 | 58 | In this study |
| R: GTTCCCTGCTGCTGTTGTTA | ||||
| CamSSR08 | F: TCACCAGTCACTTTCCCTCC | (AC)10 | 58 | In this study |
| R: CCACCAAAAGGCACAATACC | ||||
| CamSSR09 | F: CATCATCCATCAAACCGTCC | (AT)10 | 58 | In this study |
| R: GAAGGCACATTGGTTCTGGG |
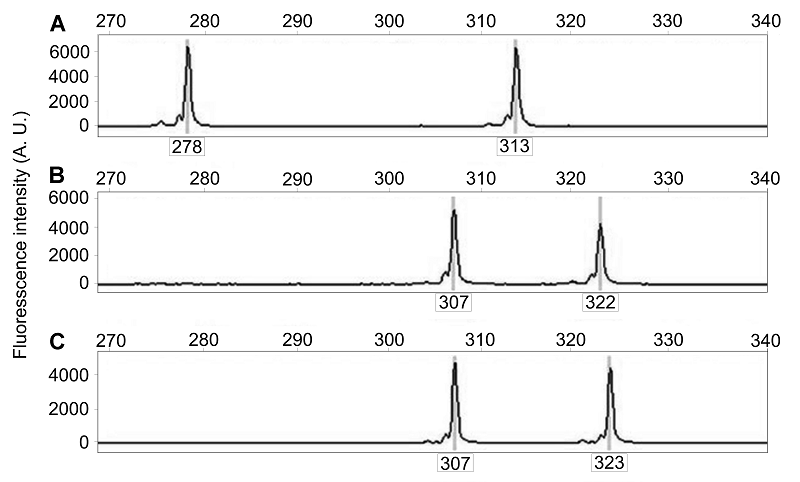
图1 3个山茶杂交新品种中荧光引物CamSSR03的SSR检测结果 (A) SJ-02品种; (B) SJ-18品种; (C) SJ-21品种
Figure 1 SSR peak map of 3 new Camellia hybrid varieties by the CamSSR03 primer (A) Variety SJ-02; (B) Variety SJ-18; (C) Variety SJ-21
| Primers | Na | Ne | PIC |
|---|---|---|---|
| 478 | 7.00 | 4.12 | 0.72 |
| SSR2 | 6.00 | 2.63 | 0.58 |
| CamsinM11 | 4.00 | 1.85 | 0.43 |
| CamSSR01 | 3.00 | 1.48 | 0.29 |
| CamSSR02 | 4.00 | 2.97 | 0.62 |
| CamSSR03 | 6.00 | 4.96 | 0.77 |
| CamSSR04 | 6.00 | 2.99 | 0.62 |
| CamSSR05 | 4.00 | 2.82 | 0.58 |
| CamSSR06 | 4.00 | 1.76 | 0.41 |
| CamSSR07 | 4.00 | 1.58 | 0.35 |
| CamSSR08 | 6.00 | 4.96 | 0.77 |
| CamSSR09 | 5.00 | 2.32 | 0.49 |
| (mean) | 4.92 | 2.87 | 0.55 |
表3 12个SSR位点的等位基因数和多态性信息
Table 3 The number of alleles and polymorphism of 12 SSR loci
| Primers | Na | Ne | PIC |
|---|---|---|---|
| 478 | 7.00 | 4.12 | 0.72 |
| SSR2 | 6.00 | 2.63 | 0.58 |
| CamsinM11 | 4.00 | 1.85 | 0.43 |
| CamSSR01 | 3.00 | 1.48 | 0.29 |
| CamSSR02 | 4.00 | 2.97 | 0.62 |
| CamSSR03 | 6.00 | 4.96 | 0.77 |
| CamSSR04 | 6.00 | 2.99 | 0.62 |
| CamSSR05 | 4.00 | 2.82 | 0.58 |
| CamSSR06 | 4.00 | 1.76 | 0.41 |
| CamSSR07 | 4.00 | 1.58 | 0.35 |
| CamSSR08 | 6.00 | 4.96 | 0.77 |
| CamSSR09 | 5.00 | 2.32 | 0.49 |
| (mean) | 4.92 | 2.87 | 0.55 |
| Code of pattern | Cam- SSR08 | Cam- SSR04 | 478 | SSR2 | Cam- SSR03 | Cam- SSR09 | Cam- SSR06 | Cam- sinM11 | Cam- SSR01 | Cam- SSR07 | Cam- SSR05 | Cam- SSR02 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 125/131 | 165/167 | 113/115 | 217 | 278/313 | 209/215 | 176/185 | 171 | 188/200 | 206 | 234/240 | 271/274 |
| 2 | 125/135 | 165/171 | 113/129 | 217/219 | 278/321 | 211/217 | 179/182 | 173 | 195 | 206/212 | 234/242 | 271/277 |
| 3 | 127 | 167 | 115 | 217/225 | 278/322 | 213/215 | 179/185 | 177 | 195/200 | 206/215 | 236/240 | 271/283 |
| 4 | 127/131 | 167/171 | 115/129 | 217/227 | 278/323 | 215 | 182/185 | 181 | 200 | 206/221 | ||
| 5 | 129 | 167/173 | 117/129 | 217/231 | 307/321 | 215/217 | 185 | |||||
| 6 | 129/131 | 169/171 | 121/129 | 217/235 | 307/322 | 217 | ||||||
| 7 | 129/133 | 171 | 123/129 | 219/235 | 307/323 | |||||||
| 8 | 129/135 | 171/173 | 123/133 | |||||||||
| 9 | 131/133 | 171/175 | ||||||||||
| A | 133 | |||||||||||
| B | 133/135 |
表4 12对引物所在位点的基因型赋值标准
Table 4 The encode standard of genotypes at 12 SSR loci in this study
| Code of pattern | Cam- SSR08 | Cam- SSR04 | 478 | SSR2 | Cam- SSR03 | Cam- SSR09 | Cam- SSR06 | Cam- sinM11 | Cam- SSR01 | Cam- SSR07 | Cam- SSR05 | Cam- SSR02 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 125/131 | 165/167 | 113/115 | 217 | 278/313 | 209/215 | 176/185 | 171 | 188/200 | 206 | 234/240 | 271/274 |
| 2 | 125/135 | 165/171 | 113/129 | 217/219 | 278/321 | 211/217 | 179/182 | 173 | 195 | 206/212 | 234/242 | 271/277 |
| 3 | 127 | 167 | 115 | 217/225 | 278/322 | 213/215 | 179/185 | 177 | 195/200 | 206/215 | 236/240 | 271/283 |
| 4 | 127/131 | 167/171 | 115/129 | 217/227 | 278/323 | 215 | 182/185 | 181 | 200 | 206/221 | ||
| 5 | 129 | 167/173 | 117/129 | 217/231 | 307/321 | 215/217 | 185 | |||||
| 6 | 129/131 | 169/171 | 121/129 | 217/235 | 307/322 | 217 | ||||||
| 7 | 129/133 | 171 | 123/129 | 219/235 | 307/323 | |||||||
| 8 | 129/135 | 171/173 | 123/133 | |||||||||
| 9 | 131/133 | 171/175 | ||||||||||
| A | 133 | |||||||||||
| B | 133/135 |
| No. | Molecular identity code | No. | Molecular identity code |
|---|---|---|---|
| SJ-01 | 192315514322 | SJ-12 | 393121414432 |
| SJ-02 | 291315514322 | SJ-13 | 485225334121 |
| SJ-03 | 122516514322 | SJ-14 | 546224111121 |
| SJ-04 | 897336114322 | SJ-15 | 545254111121 |
| SJ-05 | 975265414113 | SJ-16 | 646254114121 |
| SJ-06 | 913235513113 | SJ-17 | 353661513121 |
| SJ-07 | A73135522123 | SJ-18 | 445664323113 |
| SJ-08 | A35165424121 | SJ-19 | 934175513111 |
| SJ-09 | 973663214212 | SJ-20 | 563424544123 |
| SJ-10 | 972646514212 | SJ-21 | 748772514411 |
| SJ-11 | B61664544212 |
表6 21个山茶杂交新品种的分子身份证编码
Table 6 Molecular identity code of 21 new Camellia hybrid varieties
| No. | Molecular identity code | No. | Molecular identity code |
|---|---|---|---|
| SJ-01 | 192315514322 | SJ-12 | 393121414432 |
| SJ-02 | 291315514322 | SJ-13 | 485225334121 |
| SJ-03 | 122516514322 | SJ-14 | 546224111121 |
| SJ-04 | 897336114322 | SJ-15 | 545254111121 |
| SJ-05 | 975265414113 | SJ-16 | 646254114121 |
| SJ-06 | 913235513113 | SJ-17 | 353661513121 |
| SJ-07 | A73135522123 | SJ-18 | 445664323113 |
| SJ-08 | A35165424121 | SJ-19 | 934175513111 |
| SJ-09 | 973663214212 | SJ-20 | 563424544123 |
| SJ-10 | 972646514212 | SJ-21 | 748772514411 |
| SJ-11 | B61664544212 |
| 1 | 陈赢男, 张新叶, 戴晓港 ( 2014). 利用微卫星标记鉴别油茶品种. 经济林研究 32(4), 140-143. |
| 2 |
陈雨, 潘大建, 曲延英, 范芝兰, 陈建酉, 李晨 ( 2008). 广东高州7个普通野生稻居群遗传结构的SSR分析. 植物学报 25, 430-436.
DOI URL |
| 3 | 段云裳, 姜燕华, 王丽鸳, 成浩, 房婉萍, 黎星辉 ( 2011). 我国茶树主要骨干亲本及其衍生品种(系)的SSR分析. 植物遗传资源学报 12, 533-538. |
| 4 | 高继银, 刘信凯, 赵强民 (2016). 四季茶花杂交新品种彩色图集.杭州: 浙江科学技术出版社. pp. 26-36. |
| 5 |
郭琪, 郭大龙, 郭丽丽, 张琳, 侯小改 ( 2015). SSR分子标记在牡丹亲缘关系研究中的应用与研究进展. 植物学报 50, 652-664.
DOI URL |
| 6 |
胡兴华, 王燕, 邹玲俐, 黄仕训 ( 2013). 茶花品种SSR指纹图谱分型技术反应体系优化. 中国农学通报 29, 127-131.
DOI URL |
| 7 |
李琳琳, 黄万坚, 刘信凯, 高继银, 李凯凯, 叶创兴, 石祥刚 ( 2014). 应用SSR分子标记技术鉴定张氏红山茶杂交F1代真实性的研究. 广东园林 36(2), 44-47.
DOI URL |
| 8 |
李汝玉, 李群, 张文兰, 张晗, 宋国安, 王东建 ( 2007). 利用SSR标记进行小麦品种鉴定和新品种保护研究. 山东农业科学 ( 6), 14-17.
DOI URL |
| 9 | 李艳梅 ( 2016). 花开大理四会同办——2016大理国际茶花大会召开. 中国花卉园艺 ( 5), 36-38. |
| 10 |
刘振, 王新超, 赵丽萍, 姚明哲, 王平盛, 许玫, 唐一春, 陈亮 ( 2008). 基于EST-SSR的西南茶区茶树资源遗传多样性和亲缘关系分析. 分子植物育种 6, 100-110.
DOI URL |
| 11 | 闵天禄 (2000). 世界山茶属的研究. 昆明: 云南科技出版社. pp. 1-35. |
| 12 |
邱杨, 李锡香, 李清霞, 陈亦辰, 沈镝, 王海平, 宋江萍 ( 2014). 利用SSR标记构建萝卜种质资源分子身份证. 植物遗传资源学报 15, 648-654.
DOI URL |
| 13 | 邵阳, 范文, 黄连冬, 高继银, 李昕骥, 张文驹 ( 2015). 基于RNA-seq的崇左金花茶EST-SSR标记开发. 复旦学报(自然科学版) 54, 761-767. |
| 14 |
申屠文月, 陈析丰, 马伯军 ( 2006). 3个山茶花变异品种的形态鉴定和RAPD分析. 浙江师范大学学报(自然科学版) 29, 317-321.
DOI URL |
| 15 | 徐雷锋, 葛亮, 袁素霞, 任君芳, 袁迎迎, 李雅男, 刘春, 明军 ( 2014). 利用荧光标记SSR构建百合种质资源分子身份证. 园艺学报 41, 2055-2064. |
| 16 |
张冰清, 陆徐忠, 吴新杰, 李莉, 陈凤祥, 马琳, 张小娟, 倪金龙, 汪秀峰, 秦瑞英, 杨剑波 ( 2014). 利用SSR标记进行杂交油菜品种鉴定. 中国油料作物学报 36, 728-734.
DOI URL |
| 17 |
张嘉, 刘爱青, 张淑玲, 解莹然, 刘燕 ( 2016). 利用荧光标记SSR绘制中国芍药品种分子身份证. 北京林业大学学报 38(6), 101-109.
DOI URL |
| 18 |
张景荣, 刘军 ( 2006). 名贵茶花种质资源的RAPD分析. 西北植物学报 26, 683-687.
DOI URL |
| 19 | 张晓庆 ( 2008). 中国茶花品种分类、测试指南及已知品种数据库构建. 硕士论文. 北京: 中国林业科学研究院. pp. 33-89. |
| 20 |
张亚利, 宋垚, 奉树成 ( 2016). SSR分子标记在山茶属观赏资源遗传多样性研究中的应用. 植物科学学报 34, 755-764.
DOI URL |
| 21 | 中国国家林业局 ( 2000). 中华人民共和国林业植物新品种保护名录(第一批). 科技与法律 ( 1), 150. |
| 22 | 周文才, 温强, 杨军, 王建文, 徐立安, 徐林初 ( 2017). 油茶栽培品种SSR指纹图谱构建及聚类分析. 分子植物育种 15, 238-249. |
| 23 | Bartholomew B ( 1980). Corrigenda to the origin and classification of the garden varieties of Camellia reticulata.American Camellia Yearbook 35, 1-29. |
| 24 | Chen ZY, Jiang YS, Wang ZF, Wei JQ, Wei X, Tang H, Li ZC ( 2010). Development and characterization of microsatellite markers for Camellia nitidissima.Conserv Genet 11, 1163-1165. |
| 25 |
Cipriani G, Marrazzo MT, Marconi R, Cimato A, Testolin R ( 2002). Microsatellite markers isolated in olive ( Olea europaea L.) are suitable for individual fingerprinting and reveal polymorphism within ancient cultivars.Theor Appl Genet 104, 223-228.
DOI URL PMID |
| 26 | Doyle JJ, Doyle JL ( 1987). A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem Bull 19, 11-15. |
| 27 |
Freeman S, West J, James C, Lea V, Mayes S ( 2004). Isolation and characterization of highly polymorphic microsatellites in tea ( Camellia sinensis).Mol Ecol Notes 4, 324-326.
DOI URL |
| 28 |
Gianfranceschi L, Seglias N, Tarchini R, Komjanc M, Gessler C ( 1998). Simple sequence repeats for the ge- netic analysis of apple. Theor Appl Genet 96, 1069-1076.
DOI URL |
| 29 |
Kalia RK, Rai MK, Kalia S, Singh R, Dhawan AK ( 2011). Microsatellite markers: an overview of the recent progress in plants. Euphytica 177, 309-334.
DOI URL |
| 30 |
Kaundun SS, Matusmoto S ( 2002). Heterologous nuclear and chloroplast microsatellite amplification and variation in tea, Camellia sinensis.Genome 45, 1041-1048.
DOI URL PMID |
| 31 |
Liufu YQ, Peng GQ, Lu YB, Ye QQ, Tang SQ ( 2014). De- velopment and characterization of 38 microsatellite markers for Camellia flavida based on transcriptome sequen-cing.Conserv Genet Resour 6, 1007-1010.
DOI URL |
| 32 |
Nei M ( 1973). Analysis of gene diversity in subdivided populations. Proc Natl Acad Sci USA 70, 3321-3323.
DOI URL PMID |
| 33 |
Powell W, Machray GC, Provan J ( 1996 a). Polymorphism revealed by simple sequence repeats. Trends Plant Sci 1, 215-222.
DOI URL |
| 34 | Powell W, Morgante M, Andre C, Hanafey M, Vogel J, Tingey S, Rafalski A ( 1996 b). The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis. Mol Breed 2, 225-238. |
| 35 |
Rongwen J, Akkaya MS, Bhagwat AA, Lavi U, Cregan PB ( 1995). The use of microsatellite DNA markers for soybean genotype identification. Theor Appl Genet 90, 43-48.
DOI URL PMID |
| 36 | Wendel JF, Parks CR ( 1983). Cultivar identification in Camellia japonica L.using allozyme polymorphisms. J Amer Soc Hort Sci 108, 290-295. |
| 37 | Xu J, Huang LD, Xu Y, Zhang WJ ( 2009). Identifying hybrids of golden Camellia using SSR molecular markers.J Fudan Univ (Nat Sci) 48, 668-673. |
| 38 | Yeh FC, Yang RC, Boyle T, Ye ZH, Mao JX ( 1999). POPGENE, Version 1.32: the user friendly software for population genetic analysis. |
| [1] | 薛建华, 姜莉, 马晓林, 邴艳红, 赵思晨, 马克平. 莲品种DNA指纹图谱的构建[J]. 生物多样性, 2016, 24(1): 3-11. |
| [2] | 徐颖, 徐晶, 高继银, 张文驹. 山茶属植物ITS的多态性——一个广泛逃离一致性进化的实例[J]. 植物学报, 2011, 46(2): 162-169. |
| [3] | 梁明山 曾宇 周翔 侯留记 李霞. 遗传标记及其在作物品种鉴定中的应用[J]. 植物学报, 2001, 18(03): 257-265. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||